Résumés
Résumé
Le dépliement partiel ou le repliement alternatif d'une classe de polypeptides sont à l'origine d'événements fascinants dans la cellule : dans une conformation non native, ces polypeptides constitutifs, appelés prions, sont le vecteur d'une hérédité structurale. Ces polypeptides sont responsables d'une classe de maladies neurodégénératives chez les mammifères, et de l'apparition et la propagation de caractères phénotypiques chez la levure de boulanger. La nature du changement de conformation à l'origine de l'acquisition de propriétés infectieuses par un prion correctement replié, c'est-à-dire natif, n'est pas encore totalement connue. Les modèles mécanistiques qui peuvent rendre compte de cette hérédité structurale, ainsi que l'étendue du dépliement partiel ou du repliement alternatif des prions et leur agrégation en oligomères de masse moléculaire élevée sont présentés et discutés dans cet article. Les mécanismes potentiels de régulation de la propagation des prions par les chaperons moléculaires sont également développés.
Summary
The partial unfolding or alternative folding of a class of polypeptides is at the origin of fascinating events in living cells. In their non-native conformation, these constitutive polypeptides called prions are at the origin of a protein–based structural heredity. These polypeptides are closely associated to a class of fatal neurodegenerative illnesses in mammals and to the emergence and propagation of phenotypic traits in baker's yeasts. The structural transition from the correctly folded, native form of a prion protein to a persistent misfolded form that ultimately may cause cell death or the transmission of phenotypic traits is not yet fully understood. The mechanistic models accounting for this structure-based mode of inheritance and the extent of partial unfolding of prions or their alternative folding and the subsequent aggregation process are developed and discussed. Finally, the potential regulation of prion propagation by molecular chaperones is presented.
Corps de l’article
L'épidémie d'encéphalopathie spongiforme bovine (ou maladie de la vache folle) en Europe et le passage de l'agent infectieux chez l'homme sont à l'origine d'un fort regain d'intérêt pour les processus moléculaires à l'origine des maladies à prions.
C'est dans la seconde moitié des années 70, au Royaume-Uni, que des changements dans les procédés de valorisation de sous-produits animaux sous forme de suppléments protéiques destinés aux animaux d'élevage sont intervenus. Une nouvelle maladie, baptisée encéphalopathie spongiforme bovine (ESB) et appartenant à la famille des encéphalopathies spongiformes transmissible (EST), apparut quelques années plus tard, sans particulièrement inquiéter les autorités sanitaires dans un premier temps. En effet, une autre maladie, décrite en 1732 par des vétérinaires français et anglais et affectant des brebis qui « trottent du devant et galopent du derrière », grincent des dents et montrent une exagération des mouvements de la tête et du cou, n'avait jamais entraîné de problèmes sanitaires. Cette maladie, plus connue sous le nom de « tremblante du mouton », est transmissible au sein de l'espèce [1]. Pourtant, une relation entre la tremblante et des maladies neurodégénératives humaines comme le kuru avait été établie dès 1957 [2], et la transmissibilité interespèces démontrée [3]. Ces données eurent pour conséquence le développement de scénarios alarmants quant à la transmission de ce type de maladies de l'animal vers l'homme [4].
L'hypothèse prion
La nature non conventionnelle de l'agent infectieux de la tremblante fut documentée [5] et des hypothèses quant à son mécanisme de transmission proposées [6]. Ce n'est néanmoins qu'au début des années 80 que fut établie une relation étroite entre une protéine constitutive appelée prion, pour protéine infectieuse, et l'agent infectieux [7-11]. L'implication de la protéine prion (PrP) est restée sujette à des débats fort animés jusqu'en 1994, lorsque la démonstration fut faite qu'une souris transgénique dépourvue du gène codant pour la PrP était insensible à l'agent pathogène responsable de ces encéphalopathies transmissibles [12].
Nous ne décrirons pas ici la biosynthèse et le cycle cellulaire de la PrP, dont la fonction exacte reste inconnue. Il est néanmoins important de retenir que la PrP est une protéine dont la structure primaire est bien conservée chez les vertébrés [13], qu'elle subit des modifications post-traductionelles (N-glycosylations) [14] et qu'elle est liée à la membrane plasmique à travers une ancre glycophosphatidyl-inositol (GPI) [15-16].
Conformation de la forme infectieuse de la PrP
Des données expérimentales hétérogènes obtenues in vivo et in vitro à l'aide de méthodes indirectes ont conduit à l'idée que la forme de PrP associée à l'agent pathogène est riche en feuillets β. Parallèlement à ces travaux, la structure partielle de PrP recombinantes en solution, issues de différents vertébrés, a révélé une structure tertiaire riche en hélices α [17, 18]. Il fut alors proposé que l'acquisition du caractère infectieux de la PrP résultait d'un changement conformationnel majeur de la forme α hélicale de la PrP en une forme riche en feuillets β [19].
Cette hypothèse, encore largement admise, est régulièrement remise en question au sein de la communauté scientifique, car elle s'appuie sur une comparaison entre les propriétés de PrP recombinantes solubles, n'ayant pas subi de modifications post-traductionnelles, et celles de particules infectieuses contenant de la PrP agrégée, ayant subi des modifications post-traductionnelles et associée à des protéines qui n'ont pas toutes été identifiées à ce jour. Ces données expérimentales ont, par ailleurs, été obtenues par des mesures de dichroïsme circulaire, d'absorption dans l'infrarouge et de résistance à la protéolyse. Or le dichroïsme circulaire est une méthode inadéquate pour déterminer le contenu en structures secondaires de polypeptides agrégés [20] : en effet, même si l'activité optique dans la région 190-230 nm est dominée par le squelette d'un polypeptide, le pouvoir rotatoire d'une hélice α ou d'un feuillet β dépend de leur longueur. Cette contribution peut être estimée avec une précision acceptable lors de la décomposition des spectres expérimentaux en spectres unitaires calculés, que l'on peut comparer à ceux de différentes protéines dont la structure tridimensionnelle est connue. Toutefois, quand un polypeptide s'assemble en polymère, des éléments de structures secondaires initialement exposés au solvant se retrouvent empilés les uns au voisinage des autres. Il est alors impossible d'estimer la contribution de cet empilement au spectre de dichroïsme circulaire global, même si des spectres de protéines de structure tridimensionnelle connue sont utilisés pour exploiter les données expérimentales. Les mesures d'absorption du rayonnement infrarouge ont, quant à elles, été réalisées dans des conditions où le solvant (eau et détergent) absorbent fortement dans la région intéressante (1 600-1 700 cm-1), sur un matériel peu ou pas infectieux, et comportant un grand nombre de protéines associées à la PrP dont la contribution au signal mesuré ne peut être estimée. Enfin, la plus grande résistance du matériel infectieux à la protéolyse n'est pas une indication d'un changement de conformation de la PrP : il s'agit d'un caractère générique de tout polypeptide agrégé, riche ou non en feuillets β.
Une forme de PrP recombinante riche en brins β a été produite in vitro [21]. Cette forme n'est pas infectieuse et ne convertit pas la forme native α hélicale de la PrP en une forme non native, de façon catalytique [22] ; or, cette capacité de transformer la forme native du prion en une forme non native est à l'origine même du concept des prions. D'un autre côté, un dysfonctionnement neurologique transmissible a été rapporté lors de l'injection intracérébrale, chez des souris transgéniques surexprimant une forme tronquée de la PrP (PrP 89-231), d'une forme fibrillaire de la PrP riche en feuillets β ordonnés, dite forme amyloïde [23]. Il serait toutefois essentiel de montrer que ces observations ne sont pas dépendantes de la surexpression d'une forme tronquée de la PrP chez ces animaux, c'est-à-dire de reproduire ces résultats dans un contexte où l'animal est sauvage [24] : c'est alors, seulement, que pourrait être établi le rôle clé du changement conformationnel de la PrP et de sa forme fibrillaire dans les maladies à prion chez les mammifères.
Prions de levure
Alors que la preuve indiscutable de l'acquisition d'un caractère infectieux par la PrP à la suite d'un changement conformationnel n'est pas faite à ce jour, il existe, chez la levure de boulanger Saccharomyces cerevisiae ou chez le champignon filamenteux Podospora anserina, des protéines qui acquièrent un caractère infectieux à la suite d'un repliement alternatif.
Ces protéines ont été identifiées à la suite de l'acquisition par S. cerevisiae de phénotypes dominants, invasifs, pouvant être perdus par la cellule, mais capables de réapparaître spontanément sans introduction d'un matériel génétique étranger à la cellule (plasmides ou virus) [25-26]. Ces phénotypes, où la fonction de protéines cellulaires est perdue en l'absence de mutations dans le gène nucléaire, n'apparaissent jamais dans les cellules n'exprimant pas ces protéines. De plus, la fréquence d'apparition de ces phénotypes augmente significativement lorsque les protéines en question sont surexprimées. Ces observations ont conduit le généticien Reed Wickner à dresser un parallèle entre les propriétés imputées à la PrP et celles de ces protéines, et à proposer qu'il s'agit de prions de levure [27].
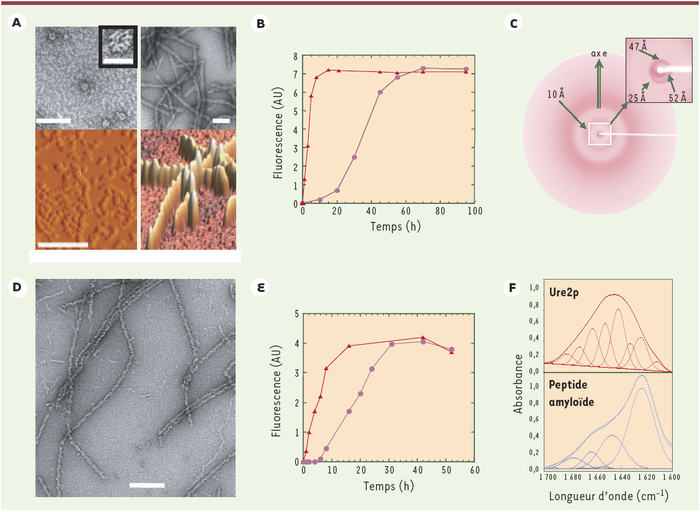
Ces protéines cytoplasmiques ont la propriété intrinsèque de s'assembler en oligomères de masse moléculaire élevée (Figure 1A et 1D). In vitro, la réaction d'assemblage de ces protéines en oligomères fibrillaires irréversibles comporte une phase de latence, étape limitante dans la réaction d'assemblage, qui peut être éliminée par l'addition de faibles quantités de fibres préformées aux solutions de protéines (Figure 1B et 1E).
Figure 1
Assemblage des prions de levure Ure2p et Sup35p en oligomères de masse moléculaire élevée et propriétés de ces oligomères.
A, D. Les formes solubles de Ure2p (A) et Sup35p (D) s'assemblent en oligomères globulaires qui s'empilent ou s'allongent pour donner naissance à des fibres. Ces différents oligomères sont observés ici par microscopie électronique à transmission et microscopie à forces atomiques (bas du panneau A) (les barres correspondent à 0,2µm en A, 0,1 µm en D et 0,02 µm dans l'insert en A). B, E. L'assemblage de ces prions comporte un temps de latence (●) que l'on peut éliminer par l'ajout de faibles quantités de fibres préformées (▲). C, F. Les clichés de diffraction des rayons X par des fibres orientées de Ure2p (C) et les spectres d'absorption du rayonnement infrarouge par ces fibres (haut du panneau F) ne comportent pas la signature des fibres amyloïdes : des réflexions à 4,7 Å et un pic d'absorption centré à 1 620cm-1 (bas du panneau F). Les fibres de Ure2p apparaissent constituées par l'empilement de molécule dans un état quasi natif, d'où les réflexions à 25 Å, 47 Å et 52 Å qui correspondent à la taille de Ure2p déterminée par cristallographie des protéines [36].
Propagation des prions
Il est très probable que les protéines prions, dans leur forme constitutive, native et active, changent de conformation pendant la phase de latence précédant leur assemblage. À la suite de ce changement de conformation, les protéines prions dans un état non natif s'associent en solution en oligomères ordonnés et stables de masse moléculaire élevée. Deux modèles de travail ont été proposés pour expliquer la propagation des formes infectieuses des protéines prions (Figure 2).
Figure 2
Modèles de propagation des prions.
Les deux modèles font appel à un changement conformationnel du prion. La forme native est en équilibre avec une forme rare, précurseur des oligomères de masse moléculaire élevée (1). A. Dans le modèle de conversion assistée, la forme non native du prion, qui peut être d'origine exogène dans les formes d'origine iatrogènes et alimentaires des maladies à prions, interagit avec la forme native du prion (2). La forme non native convertit la forme native du prion en une forme non native dans le complexe (3, 4). La dissociation de l'homodimère de forme non native produit des monomères non natifs pouvant chacun convertir la forme native du prion en forme non native (5). L'homodimère peut également croître par addition et conversion de polypeptides natifs (6). B. Dans le modèle de nucléation-polymérisation, les prions dans leur forme non native peuvent interagir les uns avec les autres longitudinalement ou latéralement (2). Ces interactions sont transitoires, car elles ne sont pas suffisamment fortes pour compenser le coût entropique de l'association. L'apparition de sites d'interaction longitudinaux et latéraux dans ces oligomères favorise fortement l'incorporation de prions dans leur forme non native, et donc la croissance et la stabilité des oligomères (3).
Les deux modèles font appel à un changement de conformation qui peut être un événement de repliement alternatif du polypeptide ayant des propriétés de prion. Cet événement est rare, sinon toute protéine prion s'agrègerait rapidement. Des mutations déstabilisant la structure native de ces polypeptides peuvent favoriser la conversion de la forme native en une forme non native, et par conséquent la probabilité d'apparition d'oligomères de masse moléculaire élevée. La forme non native est soit en équilibre avec la forme native du polypeptide prion (c'est dans ce cas que la propagation pourra avoir lieu), soit aisément identifiable et donc dégradée ou neutralisée. Les deux modèles diffèrent par la suite.
Dans le premier modèle, dit de « conversion assistée » (Figure 2A), la conversion de la forme native du prion en forme non native nécessite l'interaction directe des deux formes de la protéine. La forme non native agit dans ce modèle comme une matrice favorisant la survenue d'un événement de repliement rare. Dans le second modèle, dit de « nucléation-polymérisation » (Figure 2B), des prions dans leur forme non native peuvent interagir longitudinalement ou latéralement avec des molécules dans la même conformation. Les oligomères en formation n'acquièrent une grande stabilité qu'à partir du moment où les molécules établissent des interactions les unes avec les autres par le biais de liaisons longitudinales et latérales. L'enrôlement des formes non natives dans les polymères entraîne un déplacement de l'équilibre entre les formes native et non native, et une tendance à la production de formes non natives. Dans ces deux modèles, les oligomères peuvent se briser ou se dissocier, avec pour conséquence une augmentation de la conversion de la forme native des protéines prions en forme non native.
Caractéristiques des oligomères de masse moléculaire élevée
Des données exhaustives ne sont malheureusement pas disponibles pour tous les prions identifiés à ce jour, en particulier pour les prions de mammifères dans leur contexte naturel, c'est-à-dire associés aux membranes.
Conformation des polypeptides
Les prions de levures, dans une forme monomérique, s'associent pour former des oligomères solubles, globulaires, de masse moléculaire élevée [28, 29] (Figure 1A et 1D). Ces oligomères semblent s'empiler ou s'allonger pour former des fibres de longueur variable, pouvant dépasser 1 µm. La conformation des prions dans ces fibres reste sujette à débats, en raison de résultats contradictoires rapportés par différents groupes.
En effet, alors que les fibres produites à partir de fragments recombinants des prions de levure, insolubles et « renaturés » à partir de corps d'inclusions, sont de nature amyloïde, les fibres obtenues par l'assemblage de la forme entière soluble, au moins dans le cas du prion Ure2p à pH neutre et dans des conditions physiologiques de salinité, ne possèdent pas les signatures structurales des fibres amyloïdes. La fixation de colorants hydrophobes comme le rouge Congo à ces polymères et la biréfringence en lumière polarisée qui y est associée ne peuvent être considérées comme la preuve du contenu élevé de ces fibres en feuillets β antiparallèles ; il existe, en effet, des outils plus adéquats que la fixation d'un colorant pour déterminer si un oligomère est de nature amyloïde : il s'agit d'un diagramme caractéristique de ces structures obtenu lors de la diffraction des rayons X par des fibres orientées [30] et d'une absorbance spécifique à 1 620cm-1 dans l'infrarouge [31]. Or les fibres du prion Ure2p ne possèdent pas cette signature amyloïde (Figure 1C et 1F) et apparaissent, au contraire, constituées par l'empilement régulier de molécules dans un état quasi natif [29, 32].
Mécanismes d'assemblage à l'échelle moléculaire
Que veut dire le terme quasi natif et comment un polypeptide dans cet état peut-il s'assembler en oligomères de masse moléculaire élevée ? Le terme quasi natif n'est pas employé pour signifier que le contenu global en hélices α et brins β est identique à celui de la forme native du polypeptide, mais plutôt qu'une grande partie de la structure tertiaire est dans un état natif et peut, par exemple, fixer un ligand ou assurer une fonction catalytique.
Deux scénarios peuvent êtres envisagés pour expliquer l'acquisition, par un polypeptide, de propriétés d'assemblage en oligomères de masse moléculaire élevée (Figure 3). C'est l'exposition au solvant de quelques résidus d'acides aminés hydrophobes ou capables d'établir de nombreuses liaisons hydrogène avec une région ayant les mêmes propriétés dans ce polypeptide, à la faveur d'un dépliement limité, qui constitue le premier scénario (Figure 3A). Le second scénario fait, quant à lui, appel à l'insertion d'une hélice ou d'un brin d'un polypeptide A dans un polypeptide B, toujours à la faveur d'un changement de conformation, avec déplacement de la même hélice ou brin appartenant au peptide B (Figure 3B) : cet événement est appelé processus d'échange de domaine (domain swap). Une fois que ces interactions illégitimes sont établies, elles sont difficilement réversibles et le polypeptide est dans un état quasi natif dans les oligomères de masse moléculaire élevée. L'agrégation des flagellines bactériennes est la meilleure illustration du premier scénario [33]. Celle des serpines, dont l'agrégation est à l'origine de neurodégénerescence et de démences chez l'homme, illustre quant à elle le second scénario [34].
Figure 3
Acquisition de propriétés d'assemblage par un polypeptide dont le dépliement est limité.
A. Une région (en bleu) de quelques résidus d'acides aminés hydrophobes ou capables d'établir de nombreuses liaisons hydrogène avec une région ayant les mêmes propriétés (région matérialisée par l'encoche) dans ce polypeptide est exposée au solvant à la faveur d'un dépliement limité (1). L'interaction de cette région avec une autre région du polypeptide ayant des propriétés similaires (2) est à l'origine d'une croissance illimitée des oligomères (3). B. Le dépliement partiel du polypeptide A expose au solvant une hélice α (en bleu) ou un brin β (1). Cet élément de structure secondaire peut remplacer ou déplacer le même élément dans le polypeptide B (2). Il en résulte la formation d'un dimère dont la structure secondaire globale est identique à la somme des structures secondaires des monomères : le changement conformationnel est alors difficilement mesurable par des méthodes spectroscopiques. Ce processus d'oligomérisation par échange de domaine (domain swap) peut permettre ici aussi une croissance illimitée des oligomères (3).
Propriétés des agrégats de masse moléculaire élevée
Les oligomères ainsi formés sont associés, en particulier chez les vertébrés, à une dégénérescence cellulaire. La mort cellulaire est souvent présentée comme la conséquence de la toxicité de ces oligomères. D'autres hypothèses méritent notre attention : la mort cellulaire pourrait être la conséquence de la mobilisation, par les seuls prions, des processus cellulaires normalement destinés à empêcher l'agrégation de toute protéine cellulaire ; elle pourrait aussi résulter de la perte, au moins partielle à la suite de l'agrégation, d'une activité enzymatique essentielle au fonctionnement de la cellule. Il est enfin tentant d'envisager que ces processus d'agrégation se produisent naturellement dans la cellule, qui dispose d'un attirail moléculaire destiné à lutter contre ces événements : la mort cellulaire ne serait dans ce cas que le révélateur du vieillissement cellulaire, qui s'accompagne d'une diminution de la capacité des cellules à lutter efficacement contre l'agrégation des protéines.
Outils cellulaires de lutte contre l'agrégation
Les chaperons moléculaires, notamment le chaperon HSP104, jouent un rôle essentiel dans la propagation des phénotypes prions dans Saccharomyces cerevisiae [35]. Cette famille de protéines ubiquitaires constitue le principal outil cellulaire de lutte contre le dépliement partiel, le repliement alternatif et l'agrégation des polypeptides prions. Les événements moléculaires grâce auxquels les chaperons moléculaires modulent la propagation des prions n'ont pas encore été documentés in vitro.
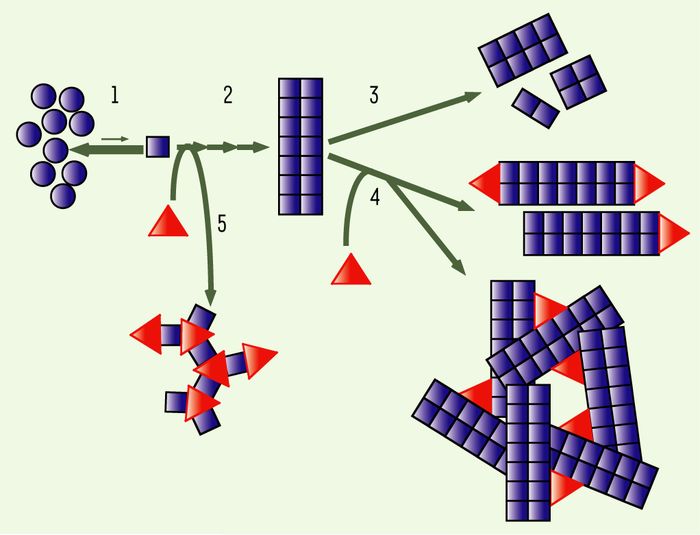
Les rôles potentiels des chaperons moléculaires et leurs différents modes d'action sont représentés dans la Figure 4. Les chaperons moléculaires pourraient désagréger ou empêcher la croissance des oligomères de masse moléculaire élevée. Dans le cas où la désagrégation serait complète, les chaperons inhiberaient la croissance des oligomères de masse moléculaire élevée. Mais si la désagrégation n'était pas complète, elle produirait un plus grand nombre d'agrégats capables, chacun, d'incorporer des polypeptides prions. Cela aurait pour conséquence de favoriser l'agrégation des prions.
Figure 4
Rôles potentiels des chaperons moléculaires dans l'agrégation des prions.
Les chaperons moléculaires (triangles rouges) peuvent agir sur l'équilibre entre les formes natives (ronds bleus) et non natives (carrés bleus) des prions (1) (voir texte). Ils peuvent également fragmenter les oligomères de masse moléculaire élevée (3) issus de l'assemblage des prions dans leur forme non native (2), ce qui a pour conséquence de favoriser l'assemblage en raison du plus grand nombre de sites d'élongation. Les chaperons moléculaires peuvent aussi coiffer les sites d'élongation, et donc limiter l'élongation, ou induire la formation d'agrégats de très haute masse moléculaire (4). Enfin, ils peuvent séquestrer la forme non native des prions dans un complexe chaperons moléculaires-prions dont les propriétés sont différentes d'un oligomère de masse moléculaire élevée constitué exclusivement de prions dans leur forme non native (5).
Les chaperons moléculaires pourraient aussi se lier aux régions exposées au solvant à la suite du dépliement partiel ou du repliement alternatif du polypeptide prion. Dans le cas où l'affinité des chaperons moléculaires pour la région reconnue est élevée, les précurseurs des oligomères de masse moléculaire élevée se retrouveraient séquestrés dans une forme incapable de s'oligomériser et la propagation des prions serait défavorisée. Si, en revanche, l'affinité des chaperons moléculaires pour les précurseurs des oligomères de prions est faible, on peut envisager qu'ils favorisent l'agrégation. En effet, comme les chaperons moléculaires interagissent avec la forme non native des prions, ils déplacent l'équilibre entre les formes native et non native vers cette dernière forme. Si par ailleurs l'affinité des chaperons pour les formes non natives des prions est faible, le complexe prion non natif-chaperon moléculaire a une probabilité élevée de se dissocier, libérant en solution le prion dans une forme non native qui donnera naissance à des oligomères de masse moléculaire élevée.
Enfin, les chaperons moléculaires pourraient, en s'associant aux oligomères de prions, induire la formation d'agrégats de très haute masse moléculaire. La déplétion de chaperons moléculaires qui résulterait de ce dernier scénario et leur séquestration dans une forme inactive pourrait avoir pour conséquence de ne plus permettre aux chaperons d'assurer leur fonction cellulaire, en particulier si la cellule est confrontée à un stress. Dans le cas où la cellule n'est pas jeune ou que l'expression du chaperon moléculaire impliqué n'est pas inductible, les processus apoptotiques seraient alors favorisés.
Conclusions et perspectives
Il est crucial de déterminer la nature exacte des changements conformationnels à l'origine de l'assemblage des prions en oligomères de masse moléculaire élevée, car cela devrait ouvrir la voie à la conception d'outils capables de moduler cette réaction ou de dissocier ces agrégats. Plusieurs approches expérimentales pourraient permettre d'atteindre cet objectif. La première est la détermination de la structure atomique des prions dans leur forme agrégée. Cet objectif ne peut pas être atteint par une approche classique de cristallographie ou de résonnance magnétique nucléaire des protéines, car ces particules sont hétérogènes et de masse moléculaire élevée. La tomographie par cryomicroscopie électronique est la méthode de choix pour accéder à la structure des prions dans ces oligomères. Cette méthode directe peut être associée à des méthodes indirectes comme l'identification des régions des prions dont l'exposition au solvant change au cours de la réaction d'assemblage, qui incluent les aires d'interactions entre molécules prions, par exemple par mesure des échanges hydrogène/deutérium (H/D) couplés à la spectrométrie de masse.
L'identification des protéines associées aux formes agrégées des prions constitue la deuxième priorité. Cet objectif peut être atteint par des approches protéomiques classiques, du type purification partielle des agrégats, séparation des polypeptides constituants sur gel d'électrophorèse bidimmensionel, et leur identification par spectrométrie de masse.
La dernière priorité est la caractérisation in vitro de l'interaction des formes natives et non natives des prions avec leurs partenaires respectifs, en particulier les chaperons moléculaires.
Parties annexes
Références
- 1. Cuille J, Chelle PL. Pathologie animale. La maladie dite tremblante du mouton est-elle inoculable? CR Acad Sci (Paris) 1936 ; 203 : 1552-4.
- 2. Hadlow WJ. Scrapie and Kuru. Lancet 1959 ; 2 : 289-90.
- 3. Chandler RL. Encephalopathy in mice produced by inoculation with scrapie brain material. Lancet 1961 ; 1 : 1378-9.
- 4. Gajdusek DC. Transmissible and non-transmissible amyloidoses: autocatalytic post-translational conversion of host precursor proteins to û-pleated conformations. J Neuroimmunol 1988 ; 20 : 95-110.
- 5. Alper T, Haig DA , Clarke MC. The exceptionally small size of the scrapie agent. Biochem Biophys Res Commun 1966 ; 22 : 278-84.
- 6. Griffith JS. Self-replication and scrapie. Nature 1967 ; 215 : 1043-4.
- 7. Prusiner SB, Groth DF, Cochran SP, et al. Molecular properties, partial purification, and assay by incubation period measurements of the hamster scrapie agent. Biochemistry 1980 ; 19 : 4883-91.
- 8. Prusiner SB, McKinley MP, Groth DF, et al. Scrapie agent contains a hydrophobic protein. Proc Natl Acad Sci USA 1981 ; 78 : 6675-9.
- 9. Prusiner SB, Bolton DC, Groth DF, et al. Further purification and characterization of scrapie prions. Biochemistry 1982 ; 26 : 6942-50.
- 10. Prusiner SB, McKinley MP, Bowman KA, et al. Scrapie prions aggregate to form amyloid-like birefringent rods. Cell 1983 ; 35 : 349-58.
- 11. Prusiner SB, Groth DF, Bolton DC, et al. Purification and structural studies of a major scrapie prion protein. Cell 1984 ; 38 : 127-34.
- 12. Sailer A, Bueler H, Fischer M, et al. No propagation of prions in mice devoid of PrP. Cell 1994 ; 77 : 967-8.
- 13. Schätzl HM, Da Costa M, Taylor L, et al. Prion protein gene variation among primates. J Mol Biol 1995 ; 254 : 362-74.
- 14. Endo T, Groth D, Prusiner SB, Kobata A. Diversity of oligosaccharide structures linked to asparagines of the scrapie prion protein. Biochemistry 1989 ; 28 : 8380-8.
- 15. Stahl N, Borchelt DR, Hsiao K, Prusiner SB. Scrapie prion protein contains a phosphatidylinositol glycolipid. Cell 1987 ; 51 : 229-40.
- 16. Rudd PM, Endo T, Colominas C, et al. Glycosylation differences between the normal and pathogenic prion protein isoforms. Proc Natl Acad Sci USA 1999 ; 96 : 13044-9.
- 17. Riek R, Hornemann S, Wider G, et al. NMR structure of the mouse prion protein domain PrP(121-321). Nature 1996 ; 382 : 180-2.
- 18. Billeter M, Riek R, Wider G, et al. Prion protein NMR structure and species barrier for prion diseases. Proc Natl Acad Sci USA 1997 ; 94 : 7281-5.
- 19. Huang Z, Prusiner SB, Cohen FE. Scrapie prions: a three-dimensional model of an infectious fragment. Folding Des 1996 ; 1 : 13-9.
- 20. Cantor CR, Schimmel PR. Biophysical chemistry, 12th ed. New York : WH Freeman and Co, 2001 : 409-31.
- 21. Jackson GS, Hosszu LL, Power A, et al. Reversible conversion of monomeric human prion protein between native and fibrilogenic conformations. Science 1999 ; 283 : 1935-7.
- 22. Hill AF, Antoniou M, Collinge. Protease-resistant prion protein produced in vitro lacks detectable infectivity. J Gen Virol 1999 ; 80 : 11-4.
- 23. Legname G, Baskakov IV, Nguyen HO, et al. Synthetic mammalian prions. Science 2004 ; 305 : 673-6.
- 24. Couzin J. An end to the prion debate? Don't count on it. Science 2004 ; 305 : 589.
- 25. Cox BS. PSI, a cytoplasmic suppressor of super-suppressor in yeast. Heredity 1965 ; 20 : 505-21.
- 26. Lacroute F. Non-Mendelian mutation allowing ureidosuccinic acid uptake in yeast. J Bacteriol 1971 ; 106 : 519-22.
- 27. Wickner RB. Evidence for a prion analog in S. cerevisiae: The [URE3] non-Mendelian genetic element as an altered URE2 protein. Science 1994 ; 264 : 566-9.
- 28. Serio TR, Cashikar AG, Kowal AS, et al. Nucleated conformational conversion and the replication of conformational information by a prion determinant. Science 2000 ; 289 : 1317-21.
- 29. Bousset L, Thomson NH, Radford SE, Melki R. The yeast prion Ure2p retains its native alpha-helical conformation upon assembly into protein fibrils in vitro. EMBO J 2002 ; 21 : 2903-11.
- 30. Sunde M, Serpell LC, Bartlam M, et al. Common core structure of amyloid fibrils by synchrotron X-ray diffraction. J Mol Biol 1997 ; 273 : 729-39.
- 31. Zurdo J, Guijarro JI, Dobson CM. Preparation and characterization of purified amyloid fibrils. J Am Chem Soc 2001 ; 123 : 8141-2.
- 32. Bousset L, Briki F, Doucet J, Melki R. The native-like conformation of Ure2p in fibrils assembled under physiologically relevant conditions switches to an amyloid-like conformation upon heat-treatment of the fibrils. J Struct Biol 2003 ; 141 : 132-42.
- 33. Abram D, Koffler H. In vitro formation of flagella-like filaments and other structure from flagellin. J Mol Biol 1964 ; 9 : 168-85.
- 34. Lomas DA, Carrell RW. Serpinopathies and the conformational dementias. Nat Rev Genet 2002 ; 3 : 759-68.
- 35. Tuite MF, Cox BS. Propagation of yeast prions. Nat Rev 2003 ; 4 : 878-89.
- 36. Bousset L, Belrhali H, Janin J, et al. Structure of the globular region of the prion protein Ure2 from the yeast Saccharomyces cerevisiae. Structure 2001 ; 9 :39-46.
Liste des figures
Figure 1
Assemblage des prions de levure Ure2p et Sup35p en oligomères de masse moléculaire élevée et propriétés de ces oligomères.
A, D. Les formes solubles de Ure2p (A) et Sup35p (D) s'assemblent en oligomères globulaires qui s'empilent ou s'allongent pour donner naissance à des fibres. Ces différents oligomères sont observés ici par microscopie électronique à transmission et microscopie à forces atomiques (bas du panneau A) (les barres correspondent à 0,2µm en A, 0,1 µm en D et 0,02 µm dans l'insert en A). B, E. L'assemblage de ces prions comporte un temps de latence (●) que l'on peut éliminer par l'ajout de faibles quantités de fibres préformées (▲). C, F. Les clichés de diffraction des rayons X par des fibres orientées de Ure2p (C) et les spectres d'absorption du rayonnement infrarouge par ces fibres (haut du panneau F) ne comportent pas la signature des fibres amyloïdes : des réflexions à 4,7 Å et un pic d'absorption centré à 1 620cm-1 (bas du panneau F). Les fibres de Ure2p apparaissent constituées par l'empilement de molécule dans un état quasi natif, d'où les réflexions à 25 Å, 47 Å et 52 Å qui correspondent à la taille de Ure2p déterminée par cristallographie des protéines [36].
Figure 2
Modèles de propagation des prions.
Les deux modèles font appel à un changement conformationnel du prion. La forme native est en équilibre avec une forme rare, précurseur des oligomères de masse moléculaire élevée (1). A. Dans le modèle de conversion assistée, la forme non native du prion, qui peut être d'origine exogène dans les formes d'origine iatrogènes et alimentaires des maladies à prions, interagit avec la forme native du prion (2). La forme non native convertit la forme native du prion en une forme non native dans le complexe (3, 4). La dissociation de l'homodimère de forme non native produit des monomères non natifs pouvant chacun convertir la forme native du prion en forme non native (5). L'homodimère peut également croître par addition et conversion de polypeptides natifs (6). B. Dans le modèle de nucléation-polymérisation, les prions dans leur forme non native peuvent interagir les uns avec les autres longitudinalement ou latéralement (2). Ces interactions sont transitoires, car elles ne sont pas suffisamment fortes pour compenser le coût entropique de l'association. L'apparition de sites d'interaction longitudinaux et latéraux dans ces oligomères favorise fortement l'incorporation de prions dans leur forme non native, et donc la croissance et la stabilité des oligomères (3).
Figure 3
Acquisition de propriétés d'assemblage par un polypeptide dont le dépliement est limité.
A. Une région (en bleu) de quelques résidus d'acides aminés hydrophobes ou capables d'établir de nombreuses liaisons hydrogène avec une région ayant les mêmes propriétés (région matérialisée par l'encoche) dans ce polypeptide est exposée au solvant à la faveur d'un dépliement limité (1). L'interaction de cette région avec une autre région du polypeptide ayant des propriétés similaires (2) est à l'origine d'une croissance illimitée des oligomères (3). B. Le dépliement partiel du polypeptide A expose au solvant une hélice α (en bleu) ou un brin β (1). Cet élément de structure secondaire peut remplacer ou déplacer le même élément dans le polypeptide B (2). Il en résulte la formation d'un dimère dont la structure secondaire globale est identique à la somme des structures secondaires des monomères : le changement conformationnel est alors difficilement mesurable par des méthodes spectroscopiques. Ce processus d'oligomérisation par échange de domaine (domain swap) peut permettre ici aussi une croissance illimitée des oligomères (3).
Figure 4
Rôles potentiels des chaperons moléculaires dans l'agrégation des prions.
Les chaperons moléculaires (triangles rouges) peuvent agir sur l'équilibre entre les formes natives (ronds bleus) et non natives (carrés bleus) des prions (1) (voir texte). Ils peuvent également fragmenter les oligomères de masse moléculaire élevée (3) issus de l'assemblage des prions dans leur forme non native (2), ce qui a pour conséquence de favoriser l'assemblage en raison du plus grand nombre de sites d'élongation. Les chaperons moléculaires peuvent aussi coiffer les sites d'élongation, et donc limiter l'élongation, ou induire la formation d'agrégats de très haute masse moléculaire (4). Enfin, ils peuvent séquestrer la forme non native des prions dans un complexe chaperons moléculaires-prions dont les propriétés sont différentes d'un oligomère de masse moléculaire élevée constitué exclusivement de prions dans leur forme non native (5).