Article body
L’apport de la levure à la condition humaine date au moins de l’utilisation du pain et du vin ; toutefois, depuis une dizaine d’années, avec la connaissance complète du génome de la levure Saccharomyces cerevisisae, la levure a une importance essentielle dans le développement des techniques et des connaissances postgénomiques. Avec ses quelque 6 000 gènes, cet eucaryote unicellulaire facilement manipulable sert de modèle pour l’étude du fonctionnement d’un génome eucaryote dont la complexité de fonctionnement nous intrigue encore.
La publication des travaux de P.Y. Lum et al. [1] nous offre une de ces utilisations élégantes de cet organisme modèle pour résoudre des questions touchant directement à la santé humaine. Nous pensons tous que la médecine va devenir de plus en plus « intelligente », et que, en particulier, nous connaîtrons précisément le mode d’action des médicaments que nous utilisons. La route est toutefois longue et tortueuse car la cellule n’offre pas une réponse simple à la présence d’un produit chimique nouveau dans son environnement ; l’approche de P.Y. Lum et al. a l’avantage de proposer une stratégie efficace pour déterminer quels gènes, et donc quelles protéines, interagissent avec un produit chimique exogène. Bien sûr, la question n’est pas nouvelle et, en ce qui concerne l’aspect in vivo, des approches génétiques conduites depuis des décennies chez la levure ont déjà apporté certaines informations. Toute-fois, l’intérêt de ce travail est de montrer comment l’utilisation in-telligente des données génomiques peut permettre de répondre à cette question de façon tellement efficace que 78 composés différents, couvrant une large gamme des chimiothérapies actuellement en usage, ont pu être étudiés.
Les auteurs ont exploité l’observation préalable selon laquelle une cellule hétérozygote - l’un des allèles correspondant à la destruction d’un gène important pour la réponse cellulaire au produit chimique étudié - croît moins vite qu’une cellule sauvage lorsqu’elle est exposée à ce nouvel environnement chimique [2]. Les auteurs ont ensuite mis au point des conditions de croissance permettant d’établir un différentiel de croissance entre les deux types de souches : présence ou absence d’un allèle impliqué dans la réponse au composé étudié. Cette sélection par la croissance (fitness) permet donc de trier les souches, donc les gènes, sensibles au composé étudié. Pour ce travail, plus de 3 500 souches ont été construites, et dans chacune, un gène différent était détruit (knock-out). Cette destruction du gène a été réalisée de telle façon que l’introduction d’une étiquette y était couplée, permettant ensuite de caractériser rapidement chacune des cicatrices correspondant, dans le génome, à l’absence du gène. En effet, ce tri naturel lié à la différence de vitesse de croissance doit être associé à un moyen efficace pour quantifier, dans le mélange des 3 500 souches, chacun des allèles impliqué. Pour cela, chaque souche portant une délétion d’un gène porte en plus une séquence spécifique, sorte de « code-barre » qui permettra, après amplification et marquage des séquences par PCR, de quantifier chaque allèle en utilisant des puces à ADN. Ainsi, après avoir laissé s’écouler le temps nécessaire à la production de 20 générations en présence d’un composé chimique, la population peut être quantitativement analysée et les gènes cibles de ce composé identifiés.
Certains résultats déjà connus, en particulier par des approches génétiques, ont été confirmés et beaucoup d’observations nouvelles ont été acquises. Ainsi, par exemple, cette approche a permis de montrer que le 5-fluoro-uracile (5-FU), un composé utilisé dans le traitement des tumeurs solides, agissait sur sept gènes qui codent pour des éléments de l’exosome et contrôlent la maturation des ARN ribosomiques. Cette observation renforce l’idée que le rôle antiprolifératif du 5-FU résulte de l’inhibition du métabolisme de l’ARN, ce qui n’était pas l’hypothèse dominante, et que l’addition d’uridine au cours du traitement diminue la toxicité et les effets pro-apoptotiques de ce médicament.
Un autre exemple est lié à l’étude de la molsidomine, un vasodilatateur utilisé en angiologie depuis une dizaine d’années. L’étude dans la levure a montré que le gène codant pour la lanostérol synthase (ERG7) est une cible de la molsidomine. Forts de ce résultat, les auteurs ont montré que, in vitro, la lanostérol synthase de plusieurs organismes pouvait effectivement être inhibée par la molsidomine. On savait déjà que la molsidomine diminue le taux de cholestérol chez l’homme et chez le rat ; cette étude offre une explication mécanistique de l’activité de ce médicament, ce qui, très probablement, devrait permettre d’améliorer ses performances. De nombreuses autres observations riches de perspectives médicales sont présentées dans cette étude concernant 78 composés chimiques.
Bien sûr, les auteurs eux-mêmes mentionnent que l’utilisation comme crible de la levure, et l’approche qu’ils décrivent dans l’article de Cell en particulier, ne peuvent sans doute pas permettre d’identifier les cibles de tous les composés chimiques. Certaines de ces cibles n’existent pas chez la levure, et si elles existent, des effets indirects peuvent les masquer. Toutefois, il est important de constater que ces études se font sans idée préconçue du rôle du composé, et qu’elles permettent d’identifier tout un groupe de protéines impliquées dans la fonction ciblée par la drogue, et pas seulement sa cible directe. On peut par ailleurs imaginer construire et utiliser des souches de levures plus sensibles à certains composés pour augmenter la sensibilité de la méthode. Cette approche offre des perspectives vraiment très riches. La raison essentielle, outre sa grande efficacité puisqu’elle prend en compte l’ensemble des gènes, réside sans doute dans le fait qu’elle est « non invasive » pour la cellule. En effet, la diminution de moitié du nombre de copies des gènes (haploinsuffisance) engendre des phénotypes subtils qui n’entraînent pas de nombreux effets indirects ; c’est un avantage considérable par rapport à des approches génétiques souvent fondées sur des phénotypes plus stricts. Notons pour finir que ce genre de travail a nécessité une collaboration étroite entre des équipes de recherche privées et publiques, associées à des moyens humains et financiers importants, toutes conditions qu’il est difficile de trouver dans notre pays ; c’est bien dommage, car les informations présentées dans cet article ne représentent sûrement qu’une partie des résultats obtenus et ne sont sans doute pas ceux qui vont conduire aux applications les plus fructueuses.
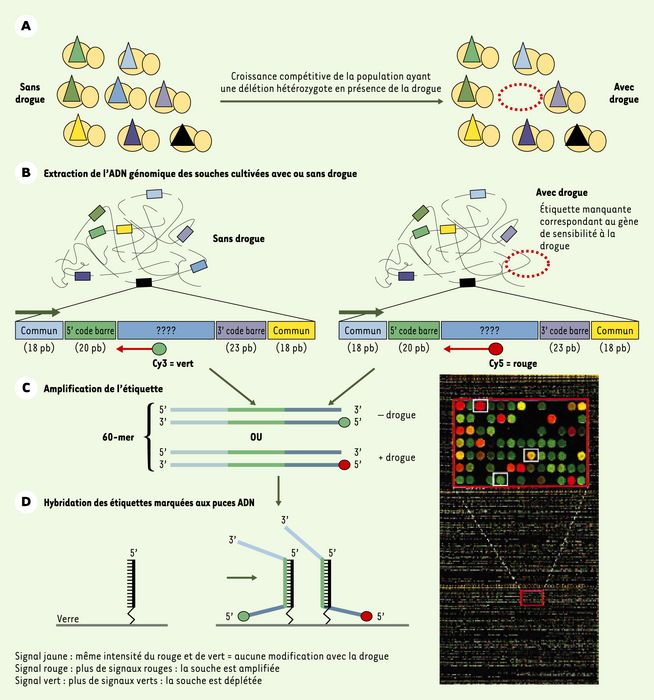
Figure 1
Stratégie pour isoler les gènes cibles d’une drogue dans une population de levure ayant des délétions hétérozygotes de gènes potentiellement cibles, ou fitness profiling.
Dans une première étape (A), les populations étiquettées et portant une délétion hétérozygote sont cultivées en l’absence ou en présence de la drogue dont on recherche les cibles. Dans une seconde étape (B), l’ADN génomique est isolé de ces cultures ; puis les « codes barres » sont amplifiés par PCR (C), marqués par les fluorochromes Cy3 ou Cy5, selon que les populations étaient cultivées en l’absence ou en présence de la drogue, et hybridés (D) sur des puces. L’intensité des signaux après hybridation est fonction de l’abondance des étiquettes dans le pool (d’après [1]).
Appendices
Références
- 1. Lum PY, Armour CD, Stepaniants SB, et al. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell 2004 ; 116 : 121-37.
- 2. Giaever G, Shoemaker DD, Jones TW, et al. Genomic profiling of drug sensitivities via induced haploinsufficiency. Nat Genet 1999 ; 21 : 278-83.
List of figures
Figure 1
Stratégie pour isoler les gènes cibles d’une drogue dans une population de levure ayant des délétions hétérozygotes de gènes potentiellement cibles, ou fitness profiling.
Dans une première étape (A), les populations étiquettées et portant une délétion hétérozygote sont cultivées en l’absence ou en présence de la drogue dont on recherche les cibles. Dans une seconde étape (B), l’ADN génomique est isolé de ces cultures ; puis les « codes barres » sont amplifiés par PCR (C), marqués par les fluorochromes Cy3 ou Cy5, selon que les populations étaient cultivées en l’absence ou en présence de la drogue, et hybridés (D) sur des puces. L’intensité des signaux après hybridation est fonction de l’abondance des étiquettes dans le pool (d’après [1]).