Corps de l’article
Les dix récentes années ont amené des percées spectaculaires dans notre compréhension de la différenciation et de la fonction de l’ostéoblaste, la cellule responsable de la formation osseuse. En particulier, l’identification et l’étude des facteurs de transcription Runx2 et Osx ont élucidé les mécanismes transcriptionnels régissant la différenciation de l’ostéoblaste et l’expression particulière des gènes qui jouent un rôle dans la fonction de ce type cellulaire [1]. Plus récemment, il est apparu que la régulation de l’activité de ces facteurs dépend notamment d’interactions protéine-protéine, comme en témoigne l’inhibition de l’activité de Runx2 par Twist‑1 et ‑2 [2]. Une compréhension globale du mécanisme de régulation transcriptionnelle de la fonction ostéoblastique appelle donc une analyse plus complète des interactions protéiques où entrent en jeu les activateurs et les répresseurs de l’expression génique dans l’ostéoblaste.
Le facteur à glissière à leucines Atf4 fait également partie des déterminants majeurs de l’activité ostéoblastique [3]. Il a été démonté qu’Atf4 module la différenciation de l’ostéoblaste [3], la transcription du gène ostéocalcine [3, 4], de même que la synthèse du collagène de type I [3]. Il est évident qu’avec un tel éventail d’actions, l’activité d’Atf4 se doit d’être sous étroite surveillance. Ainsi, la demi-vie d’Atf4 est régulée par ubiquitinylation [5]. De même, son activité transcriptionnelle est modulée après sa phosphorylation par les kinases Rsk2 (ribosomal S6 kinase-2) [3] ou PKA (protein kinase A) [6]. Nous avons identifié un mécanisme additionnel de régulation de la transcription par Atf4 : son interaction avec la protéine FIAT (factor inhibiting Atf4-mediated transcription) que notre équipe a récemment découverte et analysée [7].
FIAT est exprimée dans le noyau de l’ostéoblaste [7]. Bien que l’analyse de la séquence primaire de FIAT révèle trois motifs à glissière à leucines potentiels, la protéine ne contient pas de domaine basique qui permettrait sa liaison à l’ADN. Cette particularité suggérerait que FIAT pourrait peut-être former des dimères inactifs avec des partenaires éventuels, ce qui nous a incité à rechercher de tels partenaires en utilisant le système de deux hybrides chez la levure. À partir de banques d’ADN complémentaires provenant d’ostéoblastes primaires ou de lignées ostéoblastiques, nous avons identifié cinq clones indépendants codant pour Atf4, suggérant qu’Atf4 serait une cible préférentielle d’interaction avec FIAT [7]. Cette interaction fut confirmée dans des ostéoblastes qui expriment les deux protéines de façon endogène : l’immunoprécipitation de FIAT permet de co-immunoprécipiter Atf4, et vice-versa. Les deux protéines forment donc des dimères stables dans les cellules osseuses des mammifères.
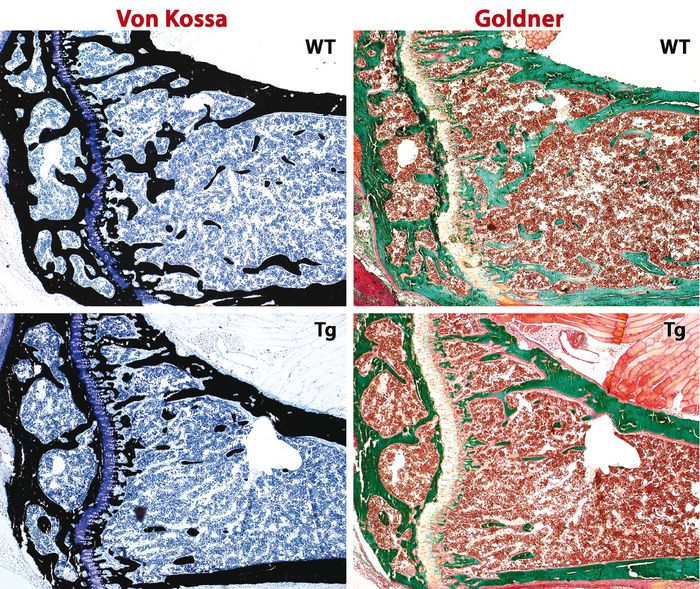
La liaison de FIAT à Atf4 inhibe l’activité transcriptionnelle d’Atf4 dans des cellules en culture. Le dimère FIAT-Atf4 ne peut se lier à l’ADN, et la transcription du gène ostéocalcine régulée par Atf4 est ainsi réduite [7]. Ce mécanisme se produit-t-il aussi in vivo ? Nous avons développé des lignées de souris transgéniques exprimant FIAT de façon exclusive dans l’ostéoblaste [7]. La transcription d’ostéocalcine est inhibée chez ces souris, et l’activité de formation osseuse des ostéoblastes est réduite. Cela conduit à une diminution significative de la masse osseuse des souris transgéniques exprimant FIAT (Figure 1), et à un affaiblissement des propriétés biomécaniques des os transgéniques. Ces changements surviennent en l’absence de perturbation de l’homéostasie des minéraux ou de changements dans la résorption osseuse [7]. Nos résultats démontrent que FIAT peut exercer une fonction décisive dans la formation osseuse in vivo et l’identifient comme un nouveau régulateur transcriptionnel de l’activité ostéoblastique.
Figure 1
Réduction de la masse osseuse chez des souris exprimant un transgène FIAT dans l’ostéoblaste.
Colorations par von Kossa (à gauche) et Goldner (à droite) de sections de tibia de souris sauvages (WT, wild type) et transgéniques (Tg) à l’âge de trois mois. Dans la coloration von Kossa, le minéral apparaît en noir. Dans la coloration Goldner, le minéral apparaît en vert. La diminution du nombre et de la dimension des trabécules osseux est évidente.
Le mécanisme d’action de FIAT comprend la formation de dimères inactifs FIAT-Atf4 (Figure 2A et B), tel que nous l’avons démontré [7]. Cependant, d’autres mécanismes pourraient aussi contribuer à l’activité de FIAT. Ainsi, FIAT pourrait interagir avec Atf4 lorsque celui-ci est lié à l’ADN, et recruter des répresseurs transcriptionnels au promoteur proximal des gènes cibles (Figure 2C). De plus, il faut tenir compte d’autres facteurs à glissière à leucines qui affectent la formation osseuse, tels que Fra-1 et ΔFosB [8, 9]. Il est conceptuellement envisageable que FIAT inhibe l’activité de ces facteurs, ou d’un de leurs partenaires d’interaction, tel que c-Jun, en séquestrant ces facteurs hors de leur site de liaison à l’ADN (Figure 2D). Nous poursuivons nos analyses structure-fonction et la recherche de partenaires d’interaction de FIAT afin de confirmer ou d’infirmer ces mécanismes potentiels.
Figure 2
Mécanismes d’action potentiels de FIAT.
La transcription du gène cible ostéocalcine est représentée en A. Lorsque FIAT interagit avec Atf4, un dimère inactif qui ne peut se lier à l’ADN est formé et la transcription du gène cible est inhibée (B). Un autre mécanisme potentiel comporte le recrutement de FIAT et d’autres répresseurs transcriptionels au promoteur proximal du gène cible (C). FIAT pourrait aussi séquestrer des facteurs à glissière à leucines, membres de la famille AP-1, tels que Fra-1, ΔFosB, ou c-Jun, et les empêcher d’activer la transcription de gènes cibles régulateurs de la masse osseuse (D). OSE1 : osteoblast specific element 1, site de liaison de Atf4 ; OSE2 : osteoblast specific element 2, site de liaison de Runx2 ; ARN Pol II : ARN polymérase II.
Nos travaux ouvrent la voie à une suite d’études qui seront déterminantes : l’inactivation ciblée de FIAT produit-elle l’effet miroir du transgène et conduit-elle à un gain de masse osseuse ? FIAT peut-elle interagir avec d’autres protéines à glissière à leucines ? Quel est le motif à glissière à leucines fonctionnel dans la séquence primaire de FIAT ? FIAT agit-elle exclusivement dans l’os ou son activité est-elle élargie ? Les réponses à ces questions augmenteront notre compréhension du rôle physiologique de FIAT et permettront d’évaluer le potentiel de FIAT comme cible d’intervention thérapeutique pour stimuler la formation osseuse dans les maladies métaboliques de l’os telle l’ostéoporose.
Parties annexes
Références
- 1. Harada S, Rodan GA. Control of osteoblast function and regulation of bone mass. Nature 2003 ; 423 : 349-55.
- 2. Bialek P, Kern B, Yang X, et al. A twist code determines the onset of osteoblast differentiation. Dev Cell 2004 ; 6 : 423-35.
- 3. Yang X, Matsuda K, Bialek P, et al. ATF4 is a substrate of RSK2 and an essential regulator of osteoblast biology. Implication for Coffin-Lowry syndrome. Cell 2004 ; 117 : 387-98.
- 4. Xiao G, Jiang D, Ge C, et al. Cooperative interactions between ATF4 and Runx2/Cbfa1 stimulate osteoblast-specific osteocalcin gene expression. J Biol Chem 2005 (sous presse).
- 5. Yang X, Karsenty G. ATF4, the osteoblast accumulation of which is determined post-translationally, can induce osteoblast-specific gene expression in non-osteoblastic cells. J Biol Chem 2004 ; 279 : 47109-14.
- 6. Elefteriou F, Ahn JD, Takeda S, et al. Leptin regulation of bone resorption by the sympathetic nervous system and CART. Nature 2005 ; 434 : 514-20.
- 7. Yu VW, Ambartsoumian G, Verlinden L, et al. FIAT represses ATF4-mediated transcription to regulate bone mass in transgenic mice. J Cell Biol 2005 ; 169 : 591-601.
- 8. Jochum W, David JP, Elliott C, et al. Increased bone formation and osteosclerosis in mice overexpressing the transcription factor Fra-1. Nat Med 2000 ; 6 : 980-4.
- 9. Sabatakos G, Sims NA, Chen J, et al. Overexpression of DeltaFosB transcription factor(s) increases bone formation and inhibits adipogenesis. Nat Med 2000 ; 6 : 985-90.
Liste des figures
Figure 1
Réduction de la masse osseuse chez des souris exprimant un transgène FIAT dans l’ostéoblaste.
Colorations par von Kossa (à gauche) et Goldner (à droite) de sections de tibia de souris sauvages (WT, wild type) et transgéniques (Tg) à l’âge de trois mois. Dans la coloration von Kossa, le minéral apparaît en noir. Dans la coloration Goldner, le minéral apparaît en vert. La diminution du nombre et de la dimension des trabécules osseux est évidente.
Figure 2
Mécanismes d’action potentiels de FIAT.
La transcription du gène cible ostéocalcine est représentée en A. Lorsque FIAT interagit avec Atf4, un dimère inactif qui ne peut se lier à l’ADN est formé et la transcription du gène cible est inhibée (B). Un autre mécanisme potentiel comporte le recrutement de FIAT et d’autres répresseurs transcriptionels au promoteur proximal du gène cible (C). FIAT pourrait aussi séquestrer des facteurs à glissière à leucines, membres de la famille AP-1, tels que Fra-1, ΔFosB, ou c-Jun, et les empêcher d’activer la transcription de gènes cibles régulateurs de la masse osseuse (D). OSE1 : osteoblast specific element 1, site de liaison de Atf4 ; OSE2 : osteoblast specific element 2, site de liaison de Runx2 ; ARN Pol II : ARN polymérase II.