Abstracts
Résumé
Les PNA (peptide nucleic acids) sont des molécules de synthèse, analogues des acides nucléiques, dans lesquelles le squelette phosphodiester a été remplacé par une chaîne pseudo-peptidique sur laquelle viennent se fixer les bases puriques et pyrimidiques. Du fait de cette structure, les molécules de PNA ne sont pas chargées et s’apparient aux séquences d’acides nucléiques avec une remarquable affinité et une très forte spécificité. De plus, les PNA présentent une remarquable stabilité et une excellente résistance aux protéases et aux nucléases. Depuis leur mise au point en 1991, les PNA ont été incorporées dans de nombreux protocoles d’études et de diagnostics génétiques pour la recherche de mutations, la coupure dirigée de l’ADN ou la thérapie génique. L’élaboration récente de sondes PNA pour l’identification in situ des chromosomes humains a permis d’adapter avec succès cette nouvelle technologie à l’analyse cytogénétique humaine.
Summary
The peptide nucleic acids (PNAs) constitute a remarkable new class of synthetic nucleic acids analogs, in which the sugar phosphate backbone is replaced by repeating N-(2-aminoethyl) glycine units linked by amine bonds and to which the nucleobases are fixed. This structure gives to PNAs the capacity to hybridize with high affinity and specificity to complementary RNA and DNA sequences, and a great resistance to nucleases and proteinases. Originally conceived as ligands for the study of double stranded DNA, the unique physico-chemical properties of PNAs have led to the development of a large variety of research and diagnostic assays, including antigene and antisense therapy and genome mapping. Several sensitive and robust PNA-dependent methods have been designed for modulating polymerase chain reactions, detecting genomic polymorphisms and mutations or capturing nucleic acids. Over the last few years, the use of PNAs has proven its powerful usefulness in cytogenetics for the rapid in situ identification of human chromosomes and the detection of aneuploidies. Recent studies have reported the successful use of chromosome-specific PNA probes on human lymphocytes, amniocytes, spermatozoa as well as on isolated oocytes and blastomeres. Muticolor PNA protocols have been described for the identification of several human chromosomes, indicating that PNAs could become a powerful tool for in situ chromosomal investigation.
Article body
Le développement d’oligonucléotides de synthèse est devenu un enjeu important dans le domaine des biotechnologies en raison de leur efficacité potentielle comme outils de diagnostic moléculaire ou agents thérapeutiques. Parmi toutes les nouvelles molécules élaborées, les PNA (peptide nucleic acids) constituent une famille remarquable d’analogues des acides nucléiques [1]. Au cours des dix dernières années, ces molécules chimériques ont été utilisées dans un nombre croissant de protocoles d’investigations et de diagnostics génétiques, et leur utilisation en thérapie génique a donné des résultats encourageants. Les espoirs suscités par les propriétés physicochimiques exceptionnelles de ces molécules semblent se confirmer avec leur introduction récente dans le domaine de la cytogénétique qui a ouvert la voie à de nouvelles applications diagnostiques très prometteuses.
Structures et propriétés des PNA
Les PNA ont été imaginées et développées par l’équipe danoise de Peter Nielsen [2]. Il s’agit de molécules synthétiques, analogues des acides nucléiques dans lesquelles le squelette phosphatidique est remplacé par une chaîne pseudopeptidique sur laquelle les bases puriques et pyrimidiques sont fixées par liaisons carboxyliques (Figure 1). Ce squelette de nature peptidique confère aux PNA des propriétés remarquables. Ainsi, contrairement aux molécules d’ADN ou d’ARN chargées négativement, les PNA ne sont pas chargées. En conséquence, il ne se produit pas de répulsion électrostatique lors de l’hybridation d’une molécule PNA avec une séquence complémentaire d’acides nucléiques, et de ce fait, les duplex PNA/ADN ou PNA/ARN présentent une spécificité d’appariement et de stabilité bien supérieures à celles des duplex naturels ADN/ADN ou ADN/ARN. Cela se traduit par des valeurs de température de dissociation (Tm) élevées pour les hybrides formés à partir de PNA, mais aussi par une plus grande sensibilité aux erreurs d’appariement des bases. Ainsi, un simple défaut d’appariement entre deux bases dans un duplex PNA/ADN de 15 nucléotides diminue la valeur de son Tm de 15°C, alors que la même erreur dans un duplex similaire ADN/ADN baisse la valeur du Tm de 10°C seulement [3]. Cette différence souligne la forte spécificité d’appariement des molécules PNA, y compris pour les PNA de petite taille.
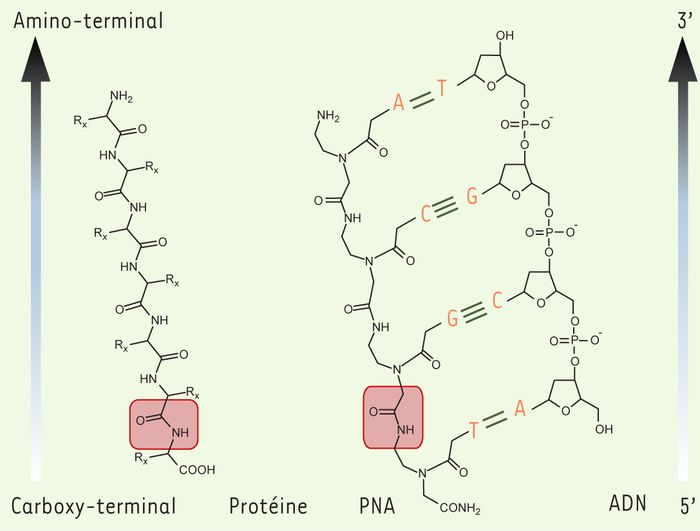
Figure 1
Structures comparées des PNA, des ADN et des protéines.
La molécule de PNA possède un squelette composé d’unités aminoéthylglycines sur lesquelles sont fixées les bases puriques et pyrimidiques, par des liaisons méthyle-carbonyle. La liaison peptidique caractéristique des PNA et des protéines est encadrée. Par convention, les molécules de PNA sont représentées comme les protéines, avec l’extrémité aminoterminale à gauche (ou en haut) et l’extrémité carboxyterminale à droite (ou en bas). Les PNA s’hybrident aux acides nucléiques selon le modèle classique de Crick et Watson d’appariement par liaisons hydrogènes (les liaisons hydrogènes sont représentées en vert).
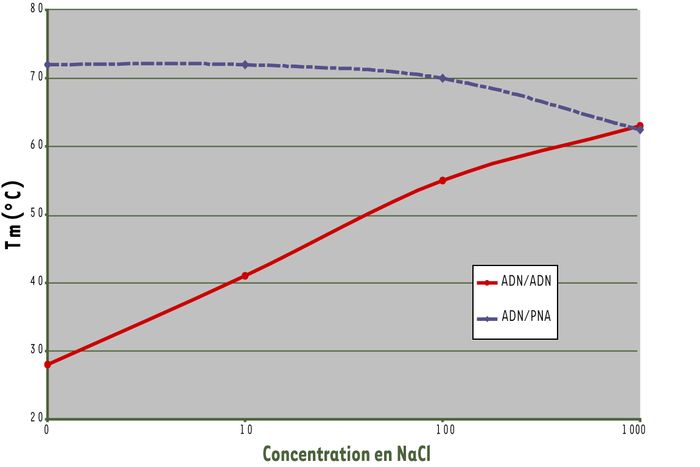
L’absence de répulsion électrostatique a pour autre conséquence une hybridation des PNA quasiment indépendante de la concentration en sel dans le milieu de réaction (Figure 2). Ainsi, pour faciliter l’accessibilité des PNA à des séquences cibles situées au coeur de structures nucléaires complexes, il est possible de déstabiliser ces structures secondaires ou tertiaires par une diminution de la concentration saline dans le milieu ambiant sans pour cela altérer l’efficacité de l’appariement des PNA [4].
Figure 2
Stabilité des duplex en fonction de la concentration en NaCl
Alors que la stabilité des hybrides formés entre deux brins d’ADN est favorisée par une force ionique élevée neutralisant les charges négatives des groupements phosphates, la concentration en sels de sodium n’a que très peu d’effet sur la stabilité des duplex PNA/ADN.
Enfin, la nature atypique du squelette des PNA permet à ces molécules de ne pas être dégradées par les nucléases ou les protéases, et de ne pas être reconnues par les polymérases. Elles ne peuvent donc pas être directement utilisées comme amorces ou être copiées in situ et présentent une très longue durée de vie in vivo et in vitro [3].
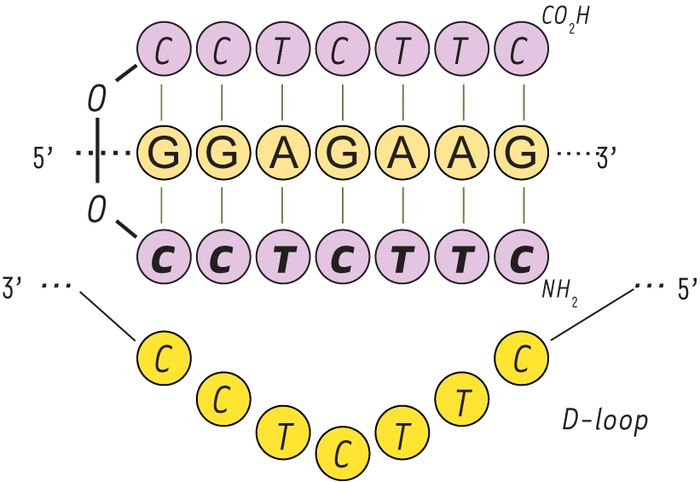
L’appariement des PNA s’effectue par complémentarité de séquences selon le modèle classique de liaisons hydrogènes de Crick et Watson, mais contrairement aux acides nucléiques, les PNA peuvent s’apparier de manière parallèle ou anti-parallèle. Les PNA s’hybrident aux acides nucléiques simple brin ou double brin. Un fait remarquable est que les PNA homopyrimidiques ou riches en bases pyrimidiques ont la capacité d’envahir la double hélice et de s’apparier à leurs séquences complémentaires selon différents schémas pouvant aboutir à la formation de structures en triple hélice (triplex) dont les plus stables, les (PNA)2/DNA, présentent des Tm très élevés [5]. Dans de tels complexes, les bases pyrimidiques des PNA forment des liaisons de type Hoogstein avec les bases puriques de l’ADN, en parallèle à l’appariement classique des bases par liaisons hydrogènes de type Crick et Watson. Ce phénomène d’invasion de la double hélice conduit à un phénomène unique en matière d’appariement qui est le déplacement d’un des deux brins d’ADN et la formation d’une boucle d’ADN simple brin (Figure 3). Ce mécanisme a été mis à profit dans plusieurs applications des PNA en biologie moléculaire.
Figure 3
Structure en triple hélice (PNA)2/DNA.
L’hybridation de deux molécules de PNA avec un brin d’ADN complémentaire entraîne le déplacement de l’autre brin d’ADN qui va ainsi former une boucle d’ADN simple brin (D-loop). Dans le triplex formé entre les deux PNA et le brin d’ADN, l’un des brins PNA est lié à l’ADN par des liaisons hydrogènes classiques de type Crick et Watson, et l’autre par des liaisons hydrogènes de type Hoogsteen (les liaisons entre les bases sont représentées par des traits verts).
Applications des PNA en génétique
Initialement conçus pour des analyses de liaison avec l’ADN, les PNA ont rapidement été exploités dans de nombreux domaines biologiques pour leur mimétisme avec les acides nucléiques et leurs propriétés physicochimiques exceptionnelles. En l’espace de dix ans, ces molécules ont été impliquées dans un nombre croissant de protocoles d’étude et de diagnostics moléculaires, confirmant ainsi l’énorme potentiel des PNA en tant qu’outils d’investigation moléculaire.
Thérapie anti-gène et anti-sens
Les premières applications des PNA ont été développées dans le domaine de la thérapie génique avec la mise au point et l’expérimentation de stratégies d’inhibition de la transcription et de la traduction des ARN [6]. Le blocage de la transcription par les PNA est fondé sur l’invasion de la double hélice et la formation de triplex au niveau du gène cible. Une sonde PNA dirigée contre la région promotrice d’un gène peut former un complexe PNA/ADN stable qui empêche le libre accès des ARN polymérases à l’ADN cible. Une hybridation intragénique de PNA peut bloquer la progression des polymérases et conduire à la production de transcrits tronqués. L’inhibition in vitro de la transcription de plusieurs gènes a ainsi été rapportée [7, 8]. Nielsen et al. [9] ont montré qu’un blocage efficace de la transcription pouvait être obtenu avec des séquences PNA inférieures à dix nucléotides. Il semble que la fixation des PNA à la double hélice soit significativement accrue en présence de superenroulements négatifs tel que ceux induits par la transcription. Paradoxalement, c’est alors le fonctionnement même de la RNA polymérase qui va entraîner l’inhibition de sa propre activité, en décuplant la fixation des molécules de PNA. On parle ainsi de « transcription suicide ». La capacité de modulation de la transcription par les PNA peut aussi être utilisée pour l’activation d’un gène cible, puisque le simple brin déplacé lors de la formation d’un triplex (PNA)2/DNA peut être reconnu par une ARN polymérase qui déclenchera une transcription à partir de ce site. Cette stratégie a été utilisée par Wang et al. [10] pour induire la transcription du gène de la gamma-globine dans des cellules de la lignée leucémique K562 où ce gène n’est habituellement pas transcrit.
Le mécanisme d’action antisens des PNA diffère de celui des oligonucléotides antisens conventionnels qui, généralement, induisent la dégradation par la RNAse H du duplex formé avec l’ARN messager. Les duplex PNA/ARN ne sont pas reconnus par la RNAse H mais agissent directement par interférences stériques au niveau de la séquence codante soit en perturbant l’attachement des ribosomes, soit en inhibant l’élongation traductionnelle. Il a également été montré que les jonctions exon-intron constituaient de bonnes cibles pour les PNA qui peuvent altérer l’épissage des ARN [11]. Dans une étude sur l’expression in vitro du gène PAL/RAR, Mologni et al. [12] ont identifié trois cibles pour les PNA antisens, à savoir les sites d’initiation AUG, la séquence codante proprement dite, et la région 5’-UTR.
Bien que ces résultats obtenus in vitro aient clairement démontré les potentialités des PNA pour la thérapie génique, l’utilisation in vivo des PNA est apparue limitée par la faible pénétration cellulaire des molécules de PNA simples, comme l’attestent les expérimentations animales d’injections cérébrales de PNA antisens [13, 14]. La pénétration intracellulaire des molécules de PNA apparaît être directement liée aux types de cellules cibles et à leurs capacités d’absorption par endocytose. Les cellules neuronales s’avèrent être les cellules présentant la meilleure absorption des molécules de PNA simples, mais dans tous les cas, de fortes doses de PNA sont requises. Les solutions proposées pour améliorer le passage transmembranaire des PNA sont fondées sur leur couplage avec des molécules présentant une bonne pénétration cellulaire, tels que des oligonucléotides, des lipides ou des peptides cationiques [15, 16]. Ainsi, des PNA couplés à la protéine Antennapedia de la drosophile ont permis d’inhiber l’activité de la télomérase dans des cellules de mélanome [17]. Des PNA ont aussi été conjugués avec des hormones stéroïdes comme la dihydrotestostérone, de manière à profiter du récepteur membranaire de ces hormones pour augmenter la cinétique de perméabilité cellulaire des PNA et permettre l’inhibition de l’expression de l’oncogène myc dans des cellules de carcinomes de la prostate. Enfin, l’utilisation de liposomes comme vecteur des PNA a également été testée avec succès [18].
Interaction des PNA avec les enzymes
Les PNA ne sont pas reconnues par les DNA polymérases ou les transcriptases inverses, mais elles peuvent être utilisées indirectement pour inhiber l’activité de ces enzymes [6]. Une sonde PNA peut ainsi bloquer l’élongation d’une amorce oligonucléotidique soit par fixation sur le brin matrice, soit par compétition avec l’amorce. Les chimères PNA/ADN peuvent être reconnues par certaines polymérases et peuvent donc être utilisées dans certains réactions de PCR [19]. La forte spécificité d’appariement des sondes PNA et la stabilité des duplex PNA/ADN formés ont été utilisées pour la détection de mutations ponctuelles de séquences nucléiques. Cette stratégie associant PNA et réaction PCR (PNA-directed PCR clamping) permet d’amplifier spécifiquement des séquences mutées après que l’appariement d’une sonde PNA spécifique de la séquence cible d’une des deux amorces PCR ait bloqué l’amplification des séquences non mutées. Cette procédure de blocage sélectif de l’amplification par PCR est si efficace qu’elle permet de détecter in vitro des mutations sur un seul nucléotide. Récemment, les nouvelles techniques de détection de mutations par électrophorèse capillaire et de PCR en temps réel ont aussi intégré l’utilisation des sondes PNA [20].
En combinaison avec des méthylases et des enzymes de restriction, les PNA peuvent être également utilisées comme agents de coupure dirigée, et leurs associations avec la S1 nucléase permet de créer des sites artificiels de restriction via le processus d’invasion de la double hélice et la formation d’une boucle simple brin pouvant être dégradée par la S1 nucléase [21].
Hybridation des PNA
Les sondes PNA peuvent être utilisées dans des protocoles d’hybridation au même titre que les sondes nucléiques naturelles ou de synthèse, mais avec l’avantage d’une spécificité et d’une capacité d’appariement accrue. L’absence de charge dans le squelette phosphatidique des PNA augmente significativement leur efficacité d‘hybridation dans les procédures ou soit la sonde, soit la cible est immobilisée (comme dans les techniques d’hybridation classique de Southern et Northern). En 1996, Perry-O’Keffe et al. [22] ont décrit une technique PNA de préhybridation sur gel permettant de simplifier la technique d’hybridation de Southern. Des sondes PNA marquées sont hybridées à un échantillon d’ADN double brin, puis le mélange est soumis à une électrophorèse sur gel permettant la séparation des molécules de PNA conjuguées et non conjuguées en fonction de la présence ou de l’absence de charges électriques. D’autres techniques de séparation sur gel ont utilisé des molécules de PNA directement intégrées dans le gel. Ces nouveaux procédés d’hybridation ont conduit au développement de protocoles sophistiqués de détection de polymorphismes et de mutations fondés sur l’emploi des PNA comme biosenseurs [23]. Par ailleurs, en tirant profil de la capacité des PNA à former des complexes stables dans des milieux pauvres en sels, des techniques rapides de coupure d’acides nucléiques simple ou double brins ont été élaborées, permettant la purification rapide et efficace d’échantillons d’ADN à partir de prélèvements de sang ou d’urine [24].
Applications des PNA en cytogénétique
En raison de leurs excellentes propriétés d’hybridation et de leur grande stabilité, les sondes PNA ont été introduites récemment dans le domaine de la cytogénétique moléculaire.
Les tests réalisés sur des préparations chromosomiques ont démontré que de courtes sondes PNA de 15 nucléotides pouvaient se substituer à des sondes ADN de taille bien supérieure.
Détection des séquences télomériques
La première utilisation in situ de sondes PNA a porté sur la détection des séquences télomériques consensus. Les sondes FISH consensus ne permettant pas une estimation précise de la taille des séquences télomériques in situ, Lansdorp et al. [25] ont utilisé une courte sonde PNA couplée à la fluorescéine pour déterminer la longueur des séquences télomériques de chaque chromosome humain dans diverses lignées hématopoïétiques, démontrant ainsi la supériorité des sondes PNA sur des sondes ADN ou ARN équivalentes. La pratique de la technique PNA-FISH s’est ainsi rapidement étendue à toute une série d’études cytogénétiques portant sur les cellules cancéreuses et l’effet de l’âge sur les télomères [26].
Identification in situ des chromosomes humains
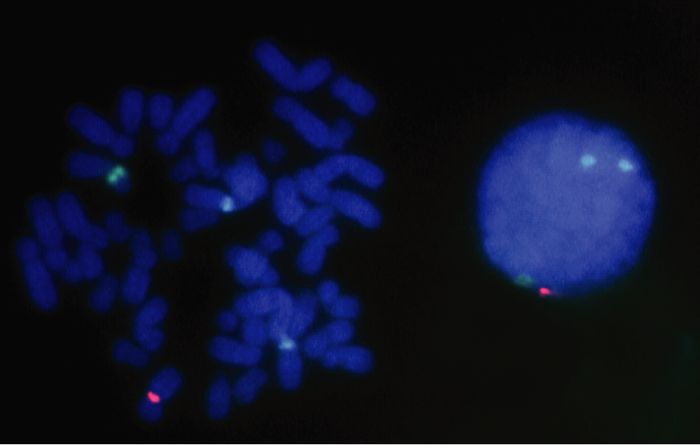
Le développement des sondes PNA s’est alors orienté vers la production de sondes pour l’identification spécifique des chromosomes humains (Figure 4). Chen et al. [27] ont élaboré les premières sondes centromériques spécifiques de chromosomes humains. La taille de ces sondes n’excédait pas 18 nucléotides et leur spécificité fut démontrée sur des préparations chromosomiques de lymphocytes et d’amniocytes normaux ou porteurs d’anomalies numériques. En 2001, Taneja et al. [28] publiaient le premier protocole de PNA-FISH multicouleur permettant le marquage de deux ou trois chromosomes en une seule étape. La forte spécificité in situ des sondes PNA a été démontrée par la mise au point de sondes permettant de distinguer des séquences d’ADN répétées présentant un seul nucléotide différent [29]. Un tel pouvoir de discrimination avait été obtenu auparavant avec la technique PRINS ou des sondes oligonucléotidiques, mais n’a jamais été atteint avec la technique de FISH conventionnelle. Aussi, la possibilité de détecter de manière aussi précise des mutations ponctuelles ou des polymorphismes in situ permet d’envisager la création de sondes PNA spécifiques d’allèle et des nouvelles applications diagnostiques.
Figure 4
Exemple de marquage PNA in situ sur une métaphase de lymphocytes humains.
La détection simultanée des chromosomes 1, X et Y a été réalisée en utilisant des sondes PNA spécifiques des centromères de chaque chromosome cible, et marquées par différents fluorochromes (chromosome 1 en bleu, chromosome X en rouge, chromosome Y en vert).
Marquage in situ des spermatozoïdes humains
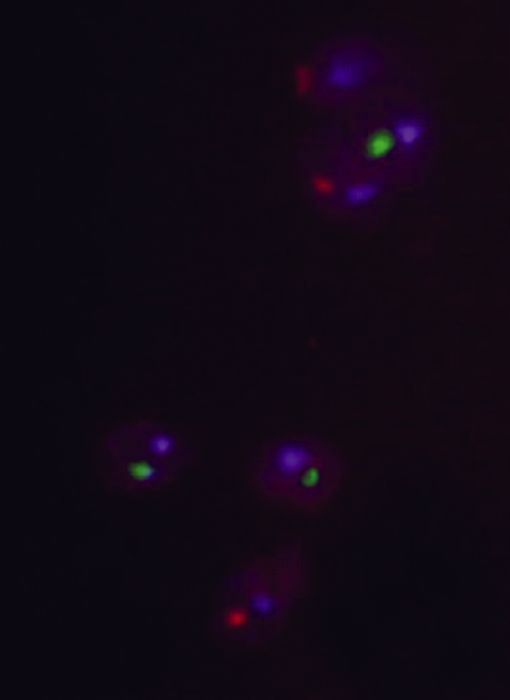
Récemment, nous avons adapté la technique PNA-FISH au marquage in situ des spermatozoïdes humains [30]. Cette adaptation constituait un test particulièrement intéressant, compte tenu de l’extrême compaction du génome des spermatozoïdes. Le marquage in situ de ces cellules nécessite une décondensation préalable de leurs noyaux et des temps d’hybridation de plusieurs heures lorsque l’on utilise des sondes ADN classiques. Les expérimentations réalisées avec les sondes PNA ont permis d’obtenir des marquages chromosomiques sur les spermatozoïdes humains dans des délais de 30 à 45 minutes (Figure 5) et des taux d’anomalies chromosomiques comparables à ceux obtenus avec les techniques FISH et PRINS.
Figure 5
Triple marquage réalisé par PNA sur un noyau de spermatozoïde humain, après décondensation de la tête spermatique par un traitement au NaOH.
Le centromère du chromosome 9 est marqué en bleu, alors que les chromosomes X et Y sont marqués respectivement en rouge et vert.
Examen cytogénétique des ovocytes et des embryons humains
Une autre application très prometteuse des sondes PNA a porté sur l’analyse chromosomique des ovocytes et des blastomères humains [31]. Le marquage simultané de plusieurs chromosomes sur ces cellules isolées a pu être réalisé en 40 minutes avec des sondes PNA spécifiques des régions centromériques des chromosomes cibles, marquées directement avec différents fluorochromes. Ces résultats ont permis de souligner la compatibilité de la technique PNA avec les impératifs de fiabilité et de rapidité exigés par le diagnostic chromosomique préimplantatoire. Dans la même optique, Agerholm et al. [32] ont récemment démontré que les sondes PNA pouvaient être combinées aux sondes ADN pour éviter les phénomènes de marquage artéfactuel sur des noyaux de blastomères humains analysés dans le cadre de diagnostics pré-implantatoires.
Conclusions
Initialement créées pour l’étude structurale de la double hélice, les sondes PNA ont progressivement été adaptées à des fins diagnostiques. Elles ont contribué à l’amélioration et à la simplification de nombreux protocoles d’investigation génétique. La mise au point d’une nouvelle génération de molécules PNA incorporant des modifications de leur squelette, devrait encore améliorer leur efficacité, et vraisemblablement réduire le coût de la synthèse des sondes PNA.
Les futures applications des PNA pourraient se développer dans des domaines où l’emploi des sondes ADN reste délicat ou inadapté. Ainsi, les courtes séquences PNA peuvent servir à l’élaboration de micropuces [33]. Leurs excellentes propriétés d’hybridation pourraient contribuer à en améliorer les procédures et les délais d’utilisation, mais aussi permettre l’utilisation des micropuces à l’échelle unicellulaire. Un autre domaine d’application des PNA pourrait être l’imagerie cellulaire in vivo. L’introduction de sondes fluorescentes dans des cellules vivantes est une approche désormais envisageable pour l’étude in vivo de l’expression génique, grâce au développement de la microscopie multiphotonique. En raison de leur grande stabilité et de leur résistance aux enzymes, les PNA apparaissent comme des outils particulièrement bien adaptés à ces nouveaux modes d’investigation cellulaire.
Appendices
Remerciements
Les auteurs remercient les laboratoires Ferring (France et Word Wide) pour le soutien apporté à ce travail. P. Paulasova bénéficie d'une bourse de recherche tchèque VZ FNM 00000064203.
Références
- 1. Saison-Behmoaras T. Les PNA : une décennie après, quels espoirs ? Med Sci (Paris) 2004 ; 20 : 148-50.
- 2. Nielsen PE, Egholm M, Berg RH, Buchardt O. Sequence-selective recognition of DNA by strand displacement with a thymine-substituted polyamide. Science 1991 ; 254 : 1497-500.
- 3. Ray A, Norden B. Peptide nucleic acid (PNA) : its medical and biotechnical applications and promise for the future. FASEB J 2000 ; 14 : 1041-60.
- 4. Gambari R. Peptide nucleic acids (PNAs) : a tool for the development of gene expression modifiers. Curr Pharma Design 2001 ; 7 : 1839-62.
- 5. Nielsen PE, Egholm M. An introduction to peptide nucleic acid. Curr Issues Mol Biol 1999 ; 1: 89-104.
- 6. Hanvey JC, Peffer NJ, Bisi JE, et al. Antisense and antigene properties of peptide nucleic acids. Science 1992 ; 258 : 1481-5.
- 7. Vickers TA, Griffity MC, Ramasamy K, et al. Inhibition of NF-kappaB specific transcriptional activation by PNA stand invasion. Nucleic Acids Res 1995 ; 23 : 3003-8.
- 8. Boffa LC, Morris PL, Carpeneto EM, et al. Invasion of the CAG triplet repeats by a complementary peptide nucleic acid inhibits transcription of the androgen receptor and TATA binding protein genes and correlates with refolding of an active nucleosome containing a unique AR gene sequence. J Biol Chem 1996 ; 271: 13228-33.
- 9. Nielsen PE, Egholm M, Buchardt O. Sequence specific transcription arrest by PNA bound to the template strand. Gene 1994 ; 149 : 139-45.
- 10. Wang G, Xu X, Pace DA, et al. Peptide nucleic acid (PNA) binding-mediated induction of human gamma-globin gene expression. Nucleic Acids Res 1999 ; 27 : 2806-13.
- 11. Doyle DF, Braasch DA, Simmons CG, et al. Inhibition of gene expression inside cells by peptide nucleic acids : effect of mRNA target sequence, mismatched bases, and PNA length. Biochemistry 2001 ; 40 : 53-64.
- 12. Mologni L, Lecoutre P, Nielsen PE, Gambacorti-Passerini C. Additive antisense effects of different PNAs on the in vitro translation of the PML/RARalpha gene. Nucleic Acids Res 1998 ; 26 : 1934-8.
- 13. Tylers BM, Jansen K, McCormick DJ, et al. Peptide nucleic acids targeted to the neurotensin receptor and administrated ip cross the blood-brain barrier and specifically reduce gene expression. Proc Natl Acad Sci USA 1999 ; 96 : 7053-8.
- 14. Fraser GL, Holmgren J, Clarke PB, Wahlesteds C. Antisense inhibition of delta-opioid receptor gene function in vivo by peptide nucleic acids. Mol Pharmacol 2000 ; 57 : 725-31.
- 15. Pooga H, Soomets U, Hällbrink M, et al. Cell penetrating PNA constructs regulate galanin receptor levels and modify pain transmission in vivo. Nat Biotechnol 1998 ; 16 : 857-61.
- 16. Cutrona G, Carpaneto EM, Ulivi M, et al. Effects in live cells of a c-myc anti-gene PNA linked to a nuclear localization signal. Nat Biotechnol 2000 ; 18 : 300-3.
- 17. Norton JC, Piatyszek MA, Wright WE, et al. Inhibition of human telomerase activity by peptide nucleic acids. Nat Biotechnol 1996 ; 14 : 615-8.
- 18. Nastruzzi C, Cortesi R, Esposito E, et al. Liposomes as carriers for DNA-PNA hybrids. J Control Release 2000 ; 10 : 237-49.
- 19. Misra HS, Pandey PK, Modak MJ, et al. Polyamide nucleic acid-DNA chimera lacking the phosphate backbone are novel primers for polymerase reaction catalyzed by DNA polymerases. Biochemistry 1998 ; 37 : 1917-25.
- 20. Isacsson J, Cao H, Ohlsson L, et al. Rapid and specific detection of PCR products using light-up probes. Mol Cell Probes 2000 ; 14 : 321-8.
- 21. Demidov V, Frank-Kamenetskii MD, Egholm M, et al. Sequence specific double strand DNA cleavage by peptide nucleic acid (PNA) targeting using nuclease SI. Nucleic Acids Res 1993 ; 21 : 2103-7.
- 22. Perry-O’Keefe H, Yao XW, Coull JM, et al. Peptide nucleic acid pre-gel hybridization : an alternative to Southern hybridization. Proc Natl Acad Sci USA 1996 ; 93 : 14670-5.
- 23. Ross PL, Lee K, Belgrader P. Discrimination of single-nucleotide polymorphisms in human DNA using peptide nucleic acid probes detected by MALDI-TOF mass spectrometry. Anal Chem 1997 ; 69 : 4197-202.
- 24. Bukanov NO, Demidov VV, Nielsen PE, Frank-Kamenetskii MD. PD-loop : a complex of duplex DNA with an oligonucleotide. Proc Natl Acad Sci USA 1998 ; 95 : 5516-20.
- 25. Lansdorp PM, Verwoerd NP, van de Rijke FM, et al. Heterogeneity in telomere length of human chromosomes. Hum Mol Genet 1996 ; 5 : 685-91.
- 26. Zijlman MJM, Martens UM, Poon SSS, et al. Telomeres in the mouse have large inter-chromosomal variations in the number of T2AG3 repeats. Proc Natl Acad Sci USA 1997 ; 94 : 7423-8.
- 27. Chen C, Wu B, Wei T, et al. Unique chromosome identification and sequence-specific structural analysis with short PNA oligomers. Mamm Genome 2000 ; 11 : 384-91.
- 28. Taneja KL, Chavez EA, Coull J, Lansdorp PM. Multicolor fluorescence in situ hybridization with peptide nucleic acid probes for enumeration of specific chromosomes in human cells. Gen Chrom Cancer 2001 ; 30 : 57-63.
- 29. Chen C, Hong YK, Ontiveros SD, et al. Single base discrimination of CENP-B repeats on mouse and human chromosomes with PNA-FISH. Mamm Genome 1999 ; 10 : 13-8.
- 30. Pellestor F, Andréo B, Taneja K, Williams B. PNA on human sperm : a new approach for in situ aneuploidy estimation. Eu J Hum Genet 2003 ; 11: 337-41.
- 31. Paulasova P, Andréo B, Diblik J, et al. The peptide nucleic acids (PNAs) as probes for chromosomal analysis : application to human oocytes, polar bodies and preimplantation embryos. Mol Hum Reprod 2004 ; 10 : 467-72.
- 32. Agerholm IE, Ziebe S, Williams B, et al. Sequential FISH analysis using competitive displacement of labelled peptide nucleic acid probes for eight chromosomes in human blastomeres. Hum Reprod 2005 ; 20 : 1072-7.
- 33. Matysiak S, Reuthner F, Hoheisel JD. Automating parallel peptide synthesis for the production of PNA library arrays. Biotechniques 2001 ; 31 : 896-904.
List of figures
Figure 1
Structures comparées des PNA, des ADN et des protéines.
La molécule de PNA possède un squelette composé d’unités aminoéthylglycines sur lesquelles sont fixées les bases puriques et pyrimidiques, par des liaisons méthyle-carbonyle. La liaison peptidique caractéristique des PNA et des protéines est encadrée. Par convention, les molécules de PNA sont représentées comme les protéines, avec l’extrémité aminoterminale à gauche (ou en haut) et l’extrémité carboxyterminale à droite (ou en bas). Les PNA s’hybrident aux acides nucléiques selon le modèle classique de Crick et Watson d’appariement par liaisons hydrogènes (les liaisons hydrogènes sont représentées en vert).
Figure 2
Stabilité des duplex en fonction de la concentration en NaCl
Figure 3
Structure en triple hélice (PNA)2/DNA.
L’hybridation de deux molécules de PNA avec un brin d’ADN complémentaire entraîne le déplacement de l’autre brin d’ADN qui va ainsi former une boucle d’ADN simple brin (D-loop). Dans le triplex formé entre les deux PNA et le brin d’ADN, l’un des brins PNA est lié à l’ADN par des liaisons hydrogènes classiques de type Crick et Watson, et l’autre par des liaisons hydrogènes de type Hoogsteen (les liaisons entre les bases sont représentées par des traits verts).
Figure 4
Exemple de marquage PNA in situ sur une métaphase de lymphocytes humains.
Figure 5
Triple marquage réalisé par PNA sur un noyau de spermatozoïde humain, après décondensation de la tête spermatique par un traitement au NaOH.