Article body
Les facteurs de transcription sterol regulatory element binding protein (SREBP) sont des acteurs majeurs de la régulation du métabolisme lipidique puisqu'ils contrôlent l'expression des gènes du métabolisme des acides gras, des triglycérides et du cholestérol [1]. Ces facteurs appartiennent à une famille composée de trois membres : SREBP-1a, SREBP-1c et SREBP-2. L'isoforme SREBP-2 est codée par le gène SREBP-2 alors que SREBP-1a et 1c sont codées par le gène SREBP-1, par l'utilisation de deux promoteurs différents et par épissage alternatif. Les trois isoformes se distinguent par leur localisation tissulaire, leurs gènes cibles et la régulation de leur activité transcriptionnelle (Tableau I) [2].
Les facteurs SREBP sont synthétisés sous la forme de précurseurs inactifs ancrés dans les membranes du réticulum endoplasmique. Le précurseur présente une structure tripartite similaire pour les trois isoformes comprenant un fragment aminoterminal correspondant au facteur de transcription, une boucle située dans la lumière du réticulum endoplasmique et un fragment carboxyterminal correspondant à un domaine de régulation. Le clivage protéolytique du précurseur SREBP permet la libération de la partie transcriptionnellement active qui, une fois libérée, migre dans le noyau pour activer ses gènes cibles [3].
Les travaux de Brown et Goldstein ont montré que la transcription et le clivage protéolytique des isoformes SREBP-1a et SREBP-2 étaient activés par la déplétion en cholestérol [1]. Pour ces isoformes, la forme mature est libérée par un processus appelé RIP (regulated intramembrane proteolysis) faisant intervenir la protéine SCAP (SREBP cleavage activating protein), les protéines Insig (insulin induced gene) et les protéases S1P et S2P [4]. La SCAP joue un double rôle : celui de cargo protéique puisqu'elle escorte le précurseur SREBP du réticulum endoplasmique vers le Golgi où ce dernier sera clivé par les deux protéases S1P et S2P, et celui de détecteur de la concentration intracellulaire de cholestérol (cholesterol sensor) puisqu'elle possède un domaine se liant au cholestérol. Ainsi, quand la concentration de cholestérol est faible, le complexe SCAP-SREBP migre du réticulum endoplasmique vers le Golgi où le précurseur est clivé par deux protéases. Au contraire, lorsque la concentration de cholestérol est élevée, le complexe SCAP-SREBP est retenu dans les membranes du réticulum endoplasmique par les protéines de rétention Insig et le précurseur n'est pas clivé (Figure 1).
Figure 1
Activation du clivage protéolytique de l'isoforme SREBP-2 par la déplétion en cholestérol.
À l'état basal, la forme précurseur SREBP-2 est ancrée dans les membranes du réticulum endoplasmique et forme un complexe avec les protéines SCAP. Le complexe SREBP-2/SCAP est retenu dans le réticulum par les protéines de rétention Insig-1. Lorsque la concentration de cholestérol diminue, la SCAP qui est un « senseur » de cholestérol change de conformation, se libère des protéines Insig et entraîne le précurseur SREBP-2 vers le Golgi par l'intermédiaire de vésicules de type COPII, où il subit un double clivage protéolytique par les protéases S1P et S2P, libérant la partie aminoterminale qui migre dans le noyau et transactive les promoteurs des gènes de biosynthèse et de capture du cholestérol.
Les études chez l'animal et dans les cultures d'hépatocytes ont montré que l'isoforme majoritaire de ces tissus, SREBP-1c, présentait une régulation différente des autres isoformes. En effet, dans des conditions de déplétion en cholestérol, alors que l'expression et les quantités de SREBP-2 nucléaires augmentent, les ARNm et la forme nucléaire de SREBP-1c ne sont que peu modifiés. Les travaux que nous avons réalisés ont permis de montrer que l'expression de l'isoforme SREBP-1c était sous le contrôle de l'environnement glucidique. L'expression de SREBP-1c est faible dans le foie d'un animal à jeun et augmente fortement lorsque celui-ci est renourri avec un régime riche en glucides. L'insuline active la transcription de SREBP-1c par la voie de la phosphatidyl inositol 3-phosphate kinase alors que le glucagon l'inhibe [5]. La surexpression de formes dominantes négatives ou positives de ce facteur a permis de démontrer que SREBP-1c était le médiateur des effets transcriptionnels positifs de l'insuline sur les gènes de la glycolyse et de la synthèse des acides gras et négatifs sur un gène clé de la néoglucogenèse [6, 7].
Le clivage protéolytique du précurseur SREBP, inactif et ancré dans les membranes du réticulum endoplasmique, est indispensable à l'induction transcriptionnelle des gènes cibles. Alors qu'il a été clairement mis en évidence que la déplétion en cholestérol est le signal qui permet de libérer les formes SREBP-2 et SREBP-1a, les mécanismes impliqués dans le clivage protéolytique de SREBP-1c restaient, jusqu'à présent, inconnus. Dans un article récent, nous montrons que le clivage protéolytique de SREBP-1c est induit très rapidement (moins de 30 minutes) en présence d'insuline dans les hépatocytes [8]. Cela se traduit par une diminution de la quantité de SREBP-1c précurseur dans le réticulum endoplasmique, et l'apparition concomitante de la forme active nucléaire (Figure 2). C'est la première fois que l'insuline est impliquée dans une régulation métabolique par l'intermédiaire d'un phénomène de protéolyse. Par quels mécanismes l'insuline stimule-t-elle la protéolyse de SREBP-1c ? Les protéines de rétention Insig pourraient jouer un rôle important dans la régulation duale des différentes isoformes par le cholestérol ou par l'insuline. Insig-1, dont l'expression est induite par l'insuline, pourrait être impliquée dans le clivage de SREBP-2 par la déplétion en cholestérol. Au contraire, Insig-2a, dont l'expression diminue en présence d'insuline, serait impliquée plus spécifiquement dans le clivage de SREBP-1c par l'insuline. L'insuline, en diminuant l'association de SCAP et d'Insig (diminution de l'expression, changement d'affinité) permettrait la libération de la forme nucléaire de SREBP-1c. Ainsi, les protéines Insig sont de bonnes candidates pour expliquer la régulation coordonnée du clivage de chacune des isoformes SREBP dans différentes conditions physiologiques.
Tableau I
Caractéristiques des trois isoformes de SREBP.
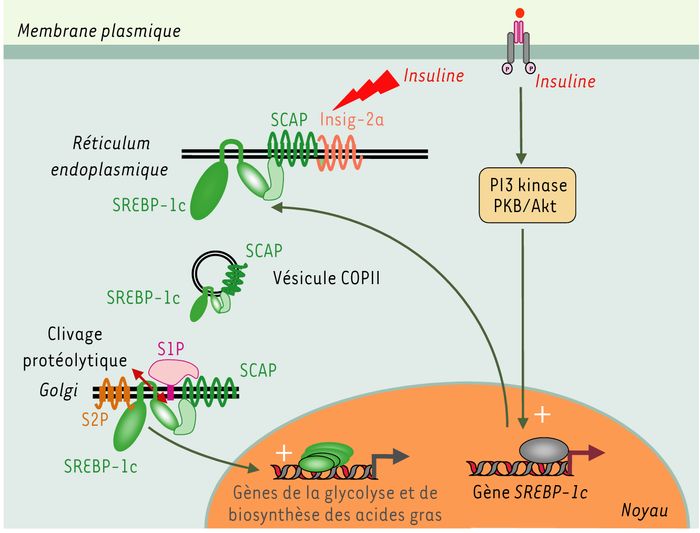
Figure 2
Activation de l'isoforme SREBP-1c par l'insuline dans le foie.
L'insuline après liaison à son récepteur active la transcription du gène SREBP-1c par une voie impliquant la phosphatidyl inositol 3 phosphate kinase (PI3 kinase) et la protéine kinase B (PKB). Cela entraîne une synthèse de la protéine précurseur de SREBP-1c qui s'ancre dans les membranes du réticulum endoplasmique. L'insuline active le clivage protéolytique de la forme précurseur de SREBP-1c. L'insuline pourrait agir en partie en diminuant l'expression de la protéine de rétention Insig-2 mais également en diminuant son affinité pour la SCAP. Cela a pour conséquence la dissociation du complexe SCAP/SREBP-1c, d'Insig. Le complexe SCAP/SREBP migre vers le Golgi par l'intermédiaire de vésicules de type COPII où il subit un double clivage protéolytique par les protéases S1P et S2P libérant la partie aminoterminale qui migre dans le noyau et transactive les promoteurs des gènes de la glycolyse et de la biosynthèse des acides gras.
Appendices
Références
- 1. Eberlé D, Hegarty B, Bossard P, et al. SREBP transcription factors: master regulators of lipid homeostasis. Biochimie 2004 ; 86 : 839-48.
- 2. Shimomura I, Shimano H, Horton JD, et al. Differential expression of exons 1and 1c in mRNAs for sterol-regulatory element binding protein-1 in human and mouse organs and cultured cells. J Clin Invest 1997 ; 99 : 838-45.
- 3. Brown MS, Goldstein JL. A proteolytic pathway that controls the cholesterol content of membranes, cells and blood. Proc Natl Acad Sci USA 1999 ; 96 : 11041-8.
- 4. Rawson RB. Control of lipid metabolism by regulated intramembrane proteolysis of sterol regulatory element binding proteins (SREBPs). Biochem Soc Symp 2003 ; 70 : 221-31
- 5. Foretz M, Pacot C, Dugail I, et al. ADD1/SREBP-1c is required in the activation of hepatic lipogenic gene expression by glucose. Mol Cell Biol 1999 ; 19 : 3760-8.
- 6. Foretz M, Guichard C, Ferré P, Foufelle F. Sterol regulatory element binding protein-1c is a major mediator of insulin action on the hepatic expression of glucokinase and lipogenesis-related genes. Proc Natl Acad Sci USA 1999 ; 96 : 12737-42.
- 7. Bécard D, Hainault I, Azzout-Marniche D, et al. Adenovirus-mediated overexpression of sterol regulatory element binding protein-1c mimics insulin effects on hepatic gene expression and glucose homeostasis in diabetic mice. Diabetes 2001 ; 50 : 2425-30.
- 8. Hegarty BD, Bobard A, Hainault, et al. Distinct roles of insulin and liver X receptor in the induction and cleavage of sterol regulatory element-binding protein-1c. Proc Natl Acad Sci USA 2005 ; 102 : 791-6.
List of figures
Figure 1
Activation du clivage protéolytique de l'isoforme SREBP-2 par la déplétion en cholestérol.
À l'état basal, la forme précurseur SREBP-2 est ancrée dans les membranes du réticulum endoplasmique et forme un complexe avec les protéines SCAP. Le complexe SREBP-2/SCAP est retenu dans le réticulum par les protéines de rétention Insig-1. Lorsque la concentration de cholestérol diminue, la SCAP qui est un « senseur » de cholestérol change de conformation, se libère des protéines Insig et entraîne le précurseur SREBP-2 vers le Golgi par l'intermédiaire de vésicules de type COPII, où il subit un double clivage protéolytique par les protéases S1P et S2P, libérant la partie aminoterminale qui migre dans le noyau et transactive les promoteurs des gènes de biosynthèse et de capture du cholestérol.
Figure 2
Activation de l'isoforme SREBP-1c par l'insuline dans le foie.
L'insuline après liaison à son récepteur active la transcription du gène SREBP-1c par une voie impliquant la phosphatidyl inositol 3 phosphate kinase (PI3 kinase) et la protéine kinase B (PKB). Cela entraîne une synthèse de la protéine précurseur de SREBP-1c qui s'ancre dans les membranes du réticulum endoplasmique. L'insuline active le clivage protéolytique de la forme précurseur de SREBP-1c. L'insuline pourrait agir en partie en diminuant l'expression de la protéine de rétention Insig-2 mais également en diminuant son affinité pour la SCAP. Cela a pour conséquence la dissociation du complexe SCAP/SREBP-1c, d'Insig. Le complexe SCAP/SREBP migre vers le Golgi par l'intermédiaire de vésicules de type COPII où il subit un double clivage protéolytique par les protéases S1P et S2P libérant la partie aminoterminale qui migre dans le noyau et transactive les promoteurs des gènes de la glycolyse et de la biosynthèse des acides gras.
List of tables
Tableau I
Caractéristiques des trois isoformes de SREBP.