Abstracts
Résumé
Les hormones thyroïdiennes (HT : T3, T4) exerçant des effets pléiotropes chez les vertébrés, leur synthèse et leur sécrétion doivent être finement contrôlées. Elles agissent elles-mêmes sur leur production, par un système de rétrocontrôle négatif de l’expression des gènes hypothalamique TRH et hypophysaire TSH. Les fondements moléculaires de cette répression transcriptionnelle des gènes TRH et TSH par l’hormone T3, forme biologiquement la plus active des HT, restent méconnus. Certaines caractéristiques de cette régulation commencent toutefois à être identifiées, notamment le rôle spécifique des isoformes TRβ (versus TRα) des récepteurs des HT. La spécificité fonctionnelle de ces isoformes résiderait principalement dans leur extrémité aminoterminale, qui permettrait une interaction différentielle avec certains comodulateurs. L’objectif, aujourd’hui, est de caractériser ces comodulateurs et d’analyser leur contribution à la régulation transcriptionnelle du gène TRH par l’hormone T3.
Summary
The synthesis and secretion of thyroid hormones (TH: T3, T4) must be strictly regulated. TH act on their own production via a negative feedback system. The synthesis of thyrotropin-releasing hormone (TRH), produced in the hypothalamus, and thyrotropin (TSH) in the pituitary is inhibited at the transcriptional level by TH. TRH and TSH stimulate production of TH. An outstanding, still open, question is the molecular basis of T3-dependent transcription repression of TRH and TSH genes. However, some regulatory components have been identified, with the β-TH receptor (TRβ) playing a specific regulatory role (versus TRα) in the negative feedback effects of T3 on production of TRH and TSH. Moreover, the N-terminus of TRβ is known to be a key element in this regulation. A hypothesis to explain this isoform specificity could be that TRβ and TRα interact differentially with transcriptional comodulators. Thus, it is critical to characterize these comodulators and to analyse their contribution to the transcription regulation of TRH.
Article body
Un des événements les plus marquants de l’évolution des vertébrés est la mise en place de contrôles endocriniens essentiels pour la régulation du développement et du métabolisme. De tels contrôles impliquent des régulations positives, par stimulation des axes hypothalamo-hypophyso-glande endocrine pour la sécrétion des hormones thyroïdiennes, des stéroïdes ou des glucocorticoïdes, puis des rétrocontrôles négatifs exercés par les hormones circulantes sur la production de facteurs hypothalamo-hypophysaires.
Les hormones thyroïdiennes (HT), tétra-iodothyronine (T4) et tri-iodothyronine (T3, forme biologiquement la plus active), sont essentielles pour le développement, la croissance et le maintien du métabolisme des vertébrés : elles influencent notamment la neurogenèse lors du développement post-embryonnaire et chez l’adulte, et contrôlent de nombreux aspects de la fonction du cerveau. De manière peut-être plus frappante encore, les HT orchestrent le contrôle de la métamorphose chez les amphibiens. Cette importance physiologique nécessite une régulation très fine de leur concentration sanguine : elles exercent elles-mêmes un rétrocontrôle négatif sur leur synthèse, au niveau des gènes codant pour des stimulateurs de leur sécrétion par la thyroïde, la TRH (thyréolibérine) dans les noyaux paraventriculaires de l’hypothalamus (PVN) et la TSH (thyréotropine) dans l’hypophyse antérieure [1-4] (Figure 1).
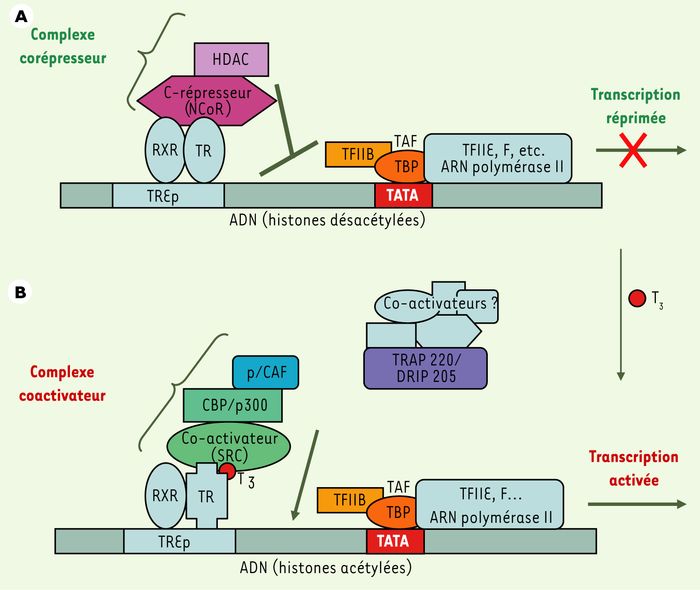
Figure 1
Mécanisme de rétrocontrôle négatif de la synthèse des hormones thyroïdiennes.
Les hormones thyroïdiennes T3 et T4 synthétisées par la thyroïde sont transportées dans le sang par des protéines plasmatiques. La T4 étant la forme majoritaire produite par la thyroïde, des désiodases vont contribuer à la désiodation de la T4 en T3, forme biologiquement active. Cette dernière agit dans les tissus périphériques en contrôlant la transcription de ses gènes cibles ; de plus, elle contrôle négativement l’expression des gènes codant pour la TSH (thyréostimuline) et la TRH (thyréolibérine), stimulatrices de la sécrétion des hormones thyroïdiennes par la thyroïde.
La T3 module l’activité transcriptionnelle de ses gènes cibles en se liant à des récepteurs nucléaires, les récepteurs des HT (TR), facteurs de transcription capables de reconnaître des séquences spécifiques (éléments de réponse aux HT, TRE) localisées le plus souvent dans les régions régulatrices des gènes cibles des HT. Selon qu’ils se fixent à des éléments de réponse positifs (TREp) ou négatifs (TREn), les TR activent ou répriment la transcription en présence de ligand. Si la plupart des travaux ont abordé les mécanismes moléculaires et les acteurs (TR, comodulateurs) impliqués dans l’activation des TREp (un modèle ayant même été établi, Figure 2), des études plus récentes ont révélé que de nombreux gènes sont également réprimés en présence d’HT, par des mécanismes encore largement méconnus. Cet article propose une synthèse des données disponibles sur les éléments de cette régulation négative, et présente un modèle de répression transcriptionnelle induite par T3 sur des TREn, la répression du gène central TRH.
Figure 2
Activation transcriptionnelle par des récepteurs d’hormones thyroïdiennes fixés sur des TRE positifs (TREp).
A. En l’absence de ligand (T3), les TR (appelés dans ce cas apo-TR) hétérodimérisés avec le facteur RXR recrutent un complexe multiprotéique corépresseur comprenant notamment des histone désacétylases (HDAC), responsables du maintien de la chromatine dans un état condensé peu favorable à la transcription. D’autres protéines interagissent également avec les cofacteurs de la machinerie transcriptionnelle de base, entraînant ainsi une répression de la transcription du gène cible. B. La liaison de la T3 aux TR (qualifiés dans ce cas d’holo-TR) induit leur changement conformationnel, permettant un relargage du complexe corépresseur au bénéfice de complexes coactivateurs comprenant des enzymes de modification des histones de type HAT (CBP/p300) et des protéines TRAP/DRIP probablement associées à d’autres coactivateurs interagissant avec la machinerie transcriptionnelle (ARNpol II + TAF + TFIIB +TBP), l’ensemble permettant une activation de la transcription (voir texte). CBP : protéine de liaison à CREB ; DRIP : protéine interagissant avec le récepteur de la vitamine D ; GTF : facteur général de transcription ; HDAC : histone désacétylase ; HAT : histone acétyltransférase ; p/CAF : facteur associé à p300/CBP ; RXR : récepteur de l’acide 9-cis-rétinoïque ; NcoR : corépresseur nucléaire ; SRC : coactivateur des récepteurs stéroïdiens ; TAF : facteur associé à TBP ; TBP : protéine de liaison à la boîte TATA ; TF : facteur de transcription ; TR : récepteur des HT ; TRAP : protéine associée aux TE ; TREp : élément de réponse positif aux TR (d’après [11]).
Activation transcriptionnelle par les HT
Comme tous les récepteurs nucléaires, les TR s’associent avec des complexes comodulateurs capables d’interagir avec la machinerie transcriptionnelle de base, ou avec les histones. La présence ou l’absence du ligand modifient la nature de ces complexes, ce qui permet de faire varier l’accessibilité de la chromatine aux facteurs généraux de transcription et de réguler la transcription génique. La Figure 2 présente les mécanismes impliqués dans la régulation transcriptionnelle par les TR fixés sur des TRE positifs.
Récepteurs des HT (TR)
Les TR, codés par deux gènes distincts NR1A1 et NR1A2 [5], partagent avec le reste de la superfamille des récepteurs nucléaires (récepteurs des stéroïdes, des rétinoïdes et de la vitamine D [6]) une organisation structurale en plusieurs domaines, dont le domaine de liaison de l’ADN (DBD, DNAbinding domain), le domaine de liaison de l’hormone (LBD, ligand binding domain) et un ou plusieurs domaines activateurs de la transcription. NR1A1 et NR1A2 codent respectivement pour cinq et quatre protéines connues à ce jour (TRα1, α2, α3, ∆α1, ∆α2 et β1, β2, β3, ∆β3), issues d’un usage alternatif de promoteur ou d’un épissage alternatif. Tous, sauf TRβ3 et ∆β3, ont été identifiés chez l’homme. Quatre seulement, TRα1, β1, β2 et α3, ont la capacité de se lier à la fois à l’hormone et à l’ADN [7-9]. Ces différentes protéines se différencient essentiellement par leurs extrémités aminoterminales, leurs DBD et LBD étant en revanche très conservés.
Chez les vertébrés adultes, TRα1 et TRβ1 ont des patrons d’expression très larges [10]. Les isoformes TRα, exprimées très tôt au cours du développement, sont retrouvées en grande quantité dans les muscles squelettiques et le tissu adipeux brun. Les isoformes TRβ présentent quant à elles une distribution spatiotemporelle plus restreinte : exprimées généralement plus tardivement au cours du développement, TRβ1 est majoritairement trouvée dans le cerveau, le foie et les reins, et TRβ2 a une distribution tissu-spécifique dans l’hypothalamus, l’hypophyse antérieure, l’oreille interne et la rétine [11, 12]. Ces variations de structure et d’expression tissulaire suggèrent fortement l’existence de différences fonctionnelles pour les différents TR.
Interactions TR/comodulateurs
D’une manière générale, les comodulateurs se liant au niveau du LBD des TR, la liaison du ligand détermine le recrutement différentiel des comodulateurs par les TR, notamment grâce à l’induction d’un changement conformationnel du récepteur. De nombreux comodulateurs pour les récepteurs nucléaires ont pu être identifiés, appartenant à la plupart des catégories fonctionnelles de protéines nucléaires.
Le premier co-activateur cloné a été SRC-1 (steroid receptor coactivator-1), identifié par une approche double-hybride sur la base de son interaction avec le récepteur de la progestérone en présence de ligand [13]. Il fut ensuite démontré que SRC-1 est également capable d’inter-agir avec les holo-TR (TR ayant fixé la T3). SRC-1 joue probablement un rôle important au sein de l’axe hypothalamo-hypophyso-thyroïdien, les souris invalidées présentant une résistance partielle aux HT. Il s’agit d’une protéine ubiquitaire d’environ 160 kDa, qui représente la famille des facteurs p160, dont la plupart possèdent une activité HAT intrinsèque, mais peuvent aussi recruter des HAT ou des méthyltransférases supplémentaires. D’autres coactivateurs de TR ont également été mis en évidence : il s’agit des familles CBP/p300 et p/CAF, et de la famille des protéines TRAP/DRIP, ces dernières assurant notamment la liaison de l’ARN polymérase II [14, 15](Figure 2B).
Quant aux corépresseurs, l’utilisation de systèmes double-hybride utilisant comme appâts le TR ou l’hétérodimère RXR/TR a montré que les protéines N-CoR [16] et SMRT (silencing mediator for retinoid and thyroid receptors) [17] interagissent avec les apo-TR (TR en l’absence de ligand), notamment sous forme de complexes avec des HDAC, et participent à la répression de la transcription de gènes régulés positivement en présence de T3(Figure 2A). Le nombre de corépresseurs et de coactivateurs des récepteurs nucléaires identifiés a considérablement augmenté, contribuant ainsi à la compréhension des mécanismes moléculaires de la régulation de nombreux gènes.
Éléments de réponse aux HT (TRE)
Les TR se lient aux TRE sous forme de monomères, d’homodimères ou d’hétérodimères avec RXR [18]. Les premiers éléments de réponse étudiés l’ont été dans le cadre d’une activation transcriptionnelle dépendante de T3 [6] : la majorité des TRE naturels caractérisés sont donc des TRE positifs (TREp) [19], formés de deux demi-sites, dont la séquence consensus est G/AGGTC/GA, et arrangés en palindromes (P), répétitions directes (DR) ou palindromes inversés (IP) espacés de 0, 4 ou 6 nucléotides, respectivement [11]. Cependant, quelques TRE négatifs (TREn) ont été également décrits, mais leurs propriétés sont bien moins connues.
Répression transcriptionnelle par les HT : le cas du gène TRH
Rôle spécifique des isoformes TR (β1/β2)
L’isoforme TRβ1 est, chez plusieurs espèces de vertébrés, majoritairement exprimée dans le cerveau, tandis que la distribution de TRβ2 se restreint notamment aux noyaux paraventriculaires de l’hypothalamus (PVN). Or ces derniers constituent également le site de production de la TRH, une production réprimée en présence de T3. Ces observations ont suggéré la possibilité que les isoformes TR (β1/β2) jouent un rôle spécifique dans la régulation transcriptionnelle négative du gène TRH par la T3. Les premiers résultats corroborant cette hypothèse ont été apportés par des études de transfection transitoire, réalisées in vitro sur des cultures primaires de neurones hypothalamiques de poulet [20], puis sur des lignées cellulaires [21, 22].
L’introduction, directement dans les hypothalamus de souris nouveau-nées, d’une construction TRH-luciférase (TRH-luc, le promoteur du gène TRH étant cloné en amont du gène rapporteur luciférase) et de vecteurs d’expression d’isoformes des TR complexés avec un polymère cationique, la polyéthylènimine, nous a permis de suivre les réponses transcriptionnelles in vivo : un des points forts de cette approche est de pouvoir modifier le statut thyroïdien des animaux et observer l’impact de ces modifications sur la transcription du gène étudié, mais aussi d’étudier les régulations physiologiques à des stades définis du développement. Les résultats montrent que le transgène TRH-luc est régulé de manière physiologique par la T3. Alors que la surexpression de TRβ1 ou TRβ2 ne perturbe pas la régulation transcriptionnelle du transgène, la surexpression de TRα1 la bloque : TRα1 semble donc incompatible avec la régulation physiologique du gène TRH, sa surexpression rendant le promoteur TRH moins accessible aux isoformes TRβ [23]. L’utilisation d’un agoniste de la T3 (GC-1) [24], qui se lie de manière préférentielle aux isoformes TRβ, nous a permis de confirmer cette hypothèse [25]. L’étude de souris n’exprimant ni TRβ1, ni TRβ2 (TRβ-/-) [26] a permis de mieux caractériser la régulation du gène TRH par la T3. Alors que le transgène TRH-luc n’est plus réprimé par la T3 chez ces animaux, le simple fait de surexprimer les isoformes TRβ1 ou TRβ2 permet de rétablir cette régulation dépendante de T3 : les isoformes TRβ sont donc toutes les deux capables d’assurer cette répression [25].
La contribution de chacune des isoformes α et β de TR a été étudiée en observant l’expression du gène TRH endogène dans les PVN de souris invalidées TRβ-/- ou TRα0/0 (gène du TRα délété) [27]. Si la répression du gène TRH dépendante de T3 est abolie chez les souris TRβ-/-, une telle répression a pu être caractérisée chez les souris TRα0/0, confirmant le rôle mineur des isoformes TRα dans ce mécanisme. Par ailleurs, une perte de répression est également observée chez les souris ayant seulement perdu l’expression de TRβ2 (TR2β-/-) [28], ce qui souligne l’importance propre de TRβ1 et de TRβ2. Enfin, les souris TRβ-/-, délétées des deux isoformes β1 et β2, présentent non seulement une perte de la répression en présence de T3, mais également une perte de l’activation en son absence [25], ce qui permet d’attribuer à TRβ1, au moins, un rôle d’activateur en l’absence de T3.
Ces expériences fonctionnelles ont soulevé la question des fondements structuraux de la spécificité de fonction des isoformes TRβ versus TRα. Les différences structurales les plus importantes résidant dans leur extrémité aminoterminale, il était possible que cette région soit responsable de la différence d’effet de TRα1 versus TRβ1/2 dans la répression transcriptionnelle du gène TRH par la T3. L’utilisation de TR chimériques, correspondant à l’échange de domaines aminoterminaux entre les deux isoformes TRα1 et TRβ1, montre que les chimères TRα1 portant l’extrémité aminoterminale de TRβ1 se comportent comme TRβ1 (de même que les chimères TRβ1 portant l’extrémité aminoterminale de TRα1 se comportent comme TRα1) lorsqu’elles sont exprimées dans l’hypothalamus de souris nouveau-nées [29]. Il semble donc que la région aminoterminale confère, à elle seule, la spécificité d’action dans la régulation négative du gène TRH [29]. Cette région serait vraisemblablement impliquée dans des interactions avec des comodulateurs nucléaires.
Rôle des complexes comodulateurs
Le rôle de différents comodulateurs dans la régulation négative du gène TRH a été étudié au sein d’un contexte hypothalamique intégré (in vivo). Le profil d’expression des deux corépresseurs SMRT et NcoR (impliqués dans la répression transcriptionnelle des gènes régulés positivement par la T3) a ainsi été analysé dans le cerveau de souris, notamment dans les PVN de l’hypothalamus, par des techniques d’hybridation in situ : l’un et l’autre sont exprimés dans les PVN, mais à des niveaux plus faibles que dans d’autres régions du cerveau (SMRT est ainsi plus fortement synthétisé dans l’hippocampe, par exemple). Cependant, même si elle est peu prononcée, leur expression s’effectue dans une zone englobant celle du gène TRH, ce qui permet de suggérer que certaines cellules à TRH co-expriment SMRT et NCoR. Une approche par transfert de gène in vivo chez la souris nouveau-née a ensuite permis de démontrer que la surexpression des corépresseurs SMRT ou NCoR entraîne une perte de la répression transcriptionnelle du transgène TRH-luc dépendante de T3 : SMRT et NCoR semblent donc interférer avec ce mécanisme plutôt qu’y contribuer, et ne sont probablement pas des partenaires privilégiés des TR dans la répression ligand-dépendante du gène TRH [30]. Cependant, des expériences d’immunoprécipitation de chromatine sur le promoteur TRH exprimé dans une lignée CV1 (qui n’exprime pas normalement ce gène) montrent un relargage de NcoR en présence de T3 [31].
En ce qui concerne les interactions TR/co-activateurs dans la régulation négative du gène TRH par la T3, il semble que les effets opposés des isoformes TRα1 et TRβ1 n’impliquent pas de différence dans le recrutement des co-activateurs, au moins en ce qui concerne SRC-1 [29]. Il est ainsi probable que d’autres cofacteurs, encore à identifier, interagissent spécifiquement avec les différents TR, entraînant des effets transcriptionnels opposés. Grâce à la technique du double-hybride chez la levure, il devrait être possible d’isoler et distinguer, à partir de banques d’ARN de PVN d’hypothalamus de souris, les partenaires préférentiels des TRβ et des TRα.
TREn et régulation du gène TRH
Quelques TREn ont été décrits, notamment ceux du gène TSHβ dans les promoteurs humain, de rat et de souris : il s’agit d’hexamères présentant une forte homologie avec les demi-sites des TREp [32-34]. Les TREn du gène TRH ont également été bien caractérisés chez l’homme [21] et chez la souris [35, 36]. Des études de mutagenèse dirigée dans le promoteur TRH humain ont permis de caractériser la région minimale de réponse transcriptionnelle à la T3, qui s’étend de - 150 à + 55 (par rapport au site d’initiation de la transcription, + 1). Dans cette région, des sites, numérotés 4, 5 et 6, sont des sites potentiels de liaison aux TR capables de participer à une répression transcriptionnelle du gène TRH dépendante de T3 [21].
Dans les analyses de transfert de gènes in vivo menées par notre équipe, la séquence promotrice du gène TRH de rat utilisée est localisée entre - 547 à + 83 : au sein de cette région, le site 4 est le seul site fonctionnel conservé chez l’homme, la souris et le rat, signant ainsi une probable importance fonctionnelle. Dans le même type d’expérience, l’utilisation d’un transgène TRH-luc dont le site 4 à été délété a permis de confirmer l’importance de ce site dans la régulation du gène TRH dépendante de T3, notamment au niveau de son activation en l’absence de ligand [29]. Les expériences de retard sur gel ont quant à elles montré l’importance du site 4 sauvage (versus muté) pour la fixation des TR. Cependant, aucune différence d’interaction de TRα1 ou TRβ1 avec le TREn du promoteur du gène TRH n’a été décelée, que ce soit en présence ou en l’absence de ligand. Au final, si le site 4 dans le promoteur TRH est bien un TREn nécessaire à la régulation du gène TRH dépendante de T3, les résultats obtenus suggèrent une fois de plus que des facteurs transcriptionnels encore inconnus interagiraient différemment avec les isoformes TRα et TRβ pour assurer leurs effets opposés.
L’analyse du promoteur du gène TRH a révélé l’existence d’un site CRE (élément de réponse à l’AMPc), juxtaposé au site 4, dont on sait qu’il participe à la régulation du promoteur du gène TRH par CREB [37]. Cette promixité des deux sites empêcherait la fixation simultanée de CREB et du TR sur leurs éléments de réponse respectifs : l’encombrement stérique créé par la liaison de CREB au site CRE pourrait alors être une composante non négligeable de la régulation du gène TRH par les TR au niveau du site 4. Ce modèle fut notamment proposé pour la régulation du gène TSH par la T3in vitro [38] : les auteurs suggéraient que le promoteur soit activé par l’AMPc en l’absence de ligand ; en présence de T3, la fixation par TRβ1 de l’histone désacétylase HDAC2, via son DBD, créerait un encombrement stérique inhibant l’activation assurée par l’AMPc.
Conclusions et perspectives
La compréhension des fondements moléculaires de la répression de la production de TRH et de TSH par les HT a une importance clinique majeure, notamment parce qu’une des complications de la thérapie des hypothyroïdies est la difficulté d’ajuster la dose de thyroxine (T4) administrée sans inhiber la production endogène de l’hormone.
Les données disponibles aujourd’hui permettent d’esquisser le schéma d’une telle régulation négative. Les récepteurs des hormones thyroïdiennes TRβ1 et TRβ2, mais pas TRα1, jouent un rôle spécifique dans la répression transcriptionnelle du gène TRH dépendante de T3, en accord avec leur niveau élevé d’expression dans l’hypothalamus. Cette spécificité des isoformes TRβ semble conférée par la nature de leur région aminoterminale, totalement divergente de celle de TRα : il est possible que TRα et TRβ adoptent des conformations distinctes sur les TREn, en lien avec cette nature distincte de leur région aminoterminale. Il semble également envisageable que des interactions amino- à carboxyterminales de certains récepteurs nucléaires puissent moduler les interactions des TR avec leurs comodulateurs, de telles interfaces TR/comodulateurs étant alors modifiées en fonction de l’élément de réponse impliqué. Des études sont en cours pour identifier les comodulateurs, exprimés dans l’hypothalamus, capables d’interagir spécifiquement avec l’une ou l’autre des isoformes du récepteur TRβ.
En conclusion, les mécanismes de répression transcriptionnelle par T3 n’ont encore jamais été modélisés, probablement en raison d’une méconnaissance des cofacteurs spécifiques impliqués. À partir de l’intégration de nos résultats et des données de la littérature, nous proposons ici un modèle de régulation négative du gène TRH dépendante de T3(Figure 3).
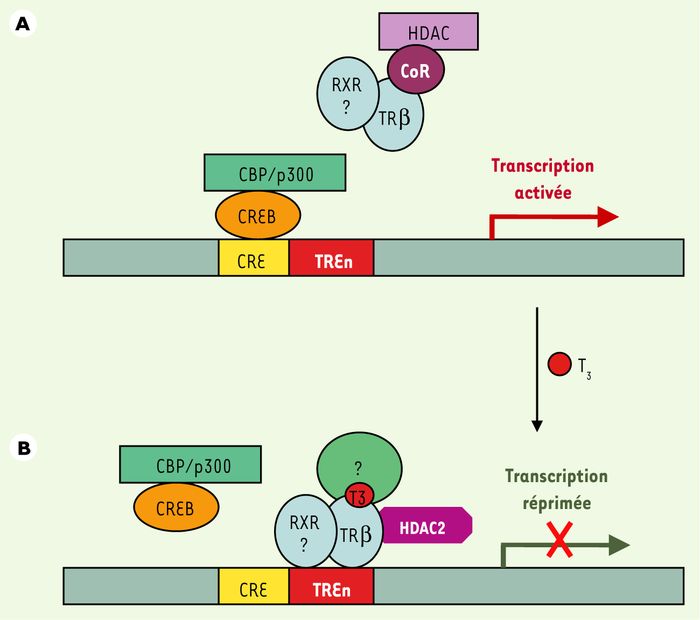
Figure 3
Modèle hypothétique de répression transcriptionnelle du gène TRH par des TRβ liés aux TRE négatifs (TREn).
A. En l’absence de T3, le gène TRH est activé par une ou plusieurs voies faisant notamment intervenir la protéine CREB et les cofacteurs qui lui sont associés. L’encombrement stérique créé par CREB et ses coactivateurs au niveau du site CRE empêcherait les TRβ de se fixer à leur TREn. Les TR, capables de recruter des complexes corépresseurs en l’absence de T3, séquestreraient ces derniers soit « en solution », soit au niveau de régions régulatrices distales du site d’initiation de la transcription. TRβ1 possède également la capacité d’induire une activation du gène TRH indépendante de T3, ce qui impliquerait peut-être la participation des facteurs cités ci-dessus, protéine CREB et coactivateurs associés [25]. B. En présence de T3, les isoformes TRβ seraient capables de se lier au TREn du gène TRH. La répression pourrait être induite via le recrutement d’une ou plusieurs protéines, peut-être spécifiques de l’hypothalamus, capables de réprimer la transcription. La possibilité que TRβ1 interagisse avec HDAC2 en présence de ligand est envisageable, cette dernière pouvant désacétyler les histones et contribuer ainsi à la répression transcriptionnelle [38]. Cela vient d’ailleurs d’être démontré dans une lignée cellulaire (qui n’exprime pas, normalement, de TRH), dans laquelle HDAC2 serait recrutée en présence de T3, provoquant la désacétylation transitoire des histones et un relargage de NCoR initialement associé à TR au niveau du promoteur TRH [31]. CRE : élément de réponse à l’AMPc ; CREB : protéine de liaison à CRE ; HDAC : histone désacétylase ; CBP/p300 : protéine de liaison à CREB ; RXR : récepteur de l’acide 9-cis-rétinoïque ; CoR : corépresseur; TR : récepteur des HT ; TREn : élément de réponse négatif aux TR.
Appendices
Références
- 1. Segerson TP, Kauer J, Wolfe HC, et al. Thyroid hormone regulates TRH biosynthesis in the paraventricular nucleus of the rat hypothalamus. Science 1987 ; 238 : 78-80.
- 2. Koller KJ, Wolff RS, Warden MK, Zoeller RT. Thyroid hormones regulate levels of thyrotropin-releasing-hormone mRNA in the paraventricular nucleus. Proc Natl Acad Sci USA 1987 ; 84 : 7329-33.
- 3. Shupnik MA, Chin WW, Ross DS, et al. Regulation by thyroxine of the mRNA encoding the alpha subunit of mouse thyrotropin. J Biol Chem 1983 ; 258 : 15120-4.
- 4. Shupnik MA, Chin WW, Ridgway EC. T3 regulation of TSH gene expression. Endocrinol Res 1989 ; 15 : 579-99.
- 5. Nuclear receptors nomenclature committee. A unified nomenclature system for the nuclear receptor superfamily. Cell 1999 ; 97 : 161-3.
- 6. Mangelsdorf DJ, Thummel C, Beato M, et al. The nuclear receptor superfamily: the second decade. Cell 1995 ; 83 : 835-9.
- 7. Lazar AL. Thyroid hormone receptors: multiple forms, multiple possibilities. Endocrine Rev 1993 ; 14 : 184-93.
- 8. Chassande O, Fraichard A, Gauthier K, et al. Identification of transcripts initiated from an internal promoter in the c-erbA alpha locus that encode inhibitors of retinoic acid receptor-alpha and triiodothyronine receptor activities. Mol Endocrinol 1997 ; 11 : 1278-90.
- 9. Williams G. Cloning and characterization of two novel thyroid hormone receptor beta isoforms. Mol Cell Biol 2000 ; 20 : 8329-42.
- 10. Hodin RA, Lazar MA, Chin WW. Differential and tissue-specific regulation of the multiple rat c-erbA messenger RNA species by thyroid hormone. J Clin Invest 1990 ; 85 : 101-5.
- 11. Yen P. Physiological and molecular basis of thyroid hormone action. Physiol Rev 2001 ; 81 : 1097-142.
- 12. Jones I, Srinivas M, Ng L, Forrest D. The thyroid hormone receptor beta gene: structure and functions in the brain and sensory systems. Thyroid 2003 ; 13 : 1057-68.
- 13. Onate SA, Tsai SY, Tsai MJ, O’Malley BW. Sequence and characterization of a coactivator for the steroid hormone receptor superfamily. Science 1995 ; 270 : 1354-7.
- 14. Chen D, Ma H, Hong H, et al. Regulation of transcription by a protein methyltransferase. Science 1999 ; 284 : 2174-7.
- 15. Rosenfeld MG, Glass CK. Coregulator codes of transcriptional regulation by nuclear receptors. J Biol Chem 2001 ; 276 : 36865-8.
- 16. Horlein AJ, Näär AM, Heinzel T, et al. Ligand-independent repression by the thyroid hormone receptor mediated by a nuclear receptor co-repressor. Nature 1995 ; 377 : 397-403.
- 17. Chen E, Evans RM. A transcriptional co-repressor that interacts with nuclear hormone receptors. Nature 1995 ; 377 : 454-7.
- 18. Mangelsdorf DJ, Evans RM. The RXR heterodimers and orphan receptors. Cell 1995 ; 83 :841-50.
- 19. Williams GR, Harney JW, Moore DD, et al. Differential capacity of wild type promoter elements for binding and trans-activation by retinoic acid and thyroid hormone receptors. Mol Endocrinol 1992 ; 6 : 1527-37.
- 20. Lezoualc’h F, Hassan AH, Giraud P, et al. Assignment of the beta-thyroid hormone receptor to 3,5,3’-triiodothyronine-dependent inhibition of transcription from the thyrotropin-releasing hormone promoter in chick hypothalamic neurons. Mol Endocrinol 1992 ; 6 : 1797-804.
- 21. Hollenberg AN, Monden T, Flynn , et al. The human thyrotropin-releasing hormone gene is regulated by thyroid hormone through two distinct classes of negative thyroid hormone response elements. Mol Endocrinol 1995 ; 9 : 540-50.
- 22. Langlois M, Zanger K, Monden T, et al. A unique role of the β-2 thyroid hormone receptor isoform in negative regulation by thyroid hormone. J Biol Chem 1997 ; 272 : 24927-33.
- 23. Guissouma H, Ghorbel TM, Seugnet I, et al. Physiological regulation of hypothalamic TRH transcription in vivo is T3 receptor isoform-specific. FASEB J 1998 ; 12 : 1755-64.
- 24. Chiellini G, Apriletti JW, Yoshihara HA, et al. A high-affinity subtype-selective agonist ligand for the thyroid hormone receptor. Chem Biol 1998 ; 5 : 299-306.
- 25. Dupre SM, Guissouma H, Flamant F, et al. Both thyroid hormone receptor (TR)beta 1 and TR beta 2 isoforms contribute to the regulation of hypothalamic thyrotropin-releasing hormone. Endocrinology 2004 ; 145 : 2337-45.
- 26. Gauthier K, Chassande O, Plateroti M, et al. Different functions for the thyroid hormone receptors TRalpha and TRbeta in the control of thyroid hormone production and post-natal development. EMBO J 1999 ; 18 : 623-31.
- 27. Gauthier K, Plateroti M, Harvey CB, et al. Genetic analysis reveals different functions for the products of the thyroid hormone receptor alpha locus. Mol Cell Biol 2001 ; 21 : 4748-60.
- 28. Abel ED, Ahima RS, Boers ME, et al. Critical role for thyroid hormone receptor beta2 in the regulation of paraventricular thyrotropin-releasing hormone neurons. J Clin Invest 2001 ; 107 : 1017-23.
- 29. Guissouma H, Dupre SM, Becker N, et al. Feedback on hypothalamic TRH transcription is dependent on thyroid hormone receptor N terminus. Mol Endocrinol 2002 ; 16 : 1652-66.
- 30. Becker N, Seugnet I, Guissouma H, et al. Nuclear corepressor and silencing mediator of retinoic and thyroid hormone receptors corepressor expression is incompatible with T(3)-dependent TRH regulation. Endocrinology 2001 ; 142 : 5321-31.
- 31. Ishii S, Yamada M, Satoh T, et al. Aberrant dynamics of histone deacetylation at the thyrotropin-releasing hormone gene in resistance to thyroid hormone. Mol Endocrinol 2004 ; 18 : 1708-20.
- 32. Wood WM, Kao MY, Gordon DF, Chester RE. Thyroid hormone regulates the mouse thyrotropin β-subunit gene promoter in transfected primary thyrotropes. J Biol Chem 1989 ; 264 : 14840-7.
- 33. Bodenner DL, Mroczynski MA, Weintraub BD, et al. A detailed functional and structural analysis of a major thyroid hormone inhibitory element in the human thyrotropin beta-subunit gene. J Biol Chem 1991 ; 266 : 21666-73.
- 34. Carr FE, Kaceem LL, Wong NC. Thyroid hormone inhibits thyrotropin gene expression via a position-independent negative L-triiodothyronine-responsive element. J Biol Chem 1992 ; 267 : 18689-94.
- 35. Satoh T, Yamada M, Iwasaki T, Mori M. Negative regulation of the gene for the preprothyrotropin-releasing hormone from the mouse by thyroid hormone requires additional factors in conjunction with thyroid hormone receptors. J Biol Chem 1996 ; 271 : 27919-26.
- 36. Satoh T, Monden T, Ishizuka T, et al. DNA binding and interaction with the nuclear receptor corepressor of thyroid hormone receptor are required for ligand-independent stimulation of the mouse preprothyrotropin-releasing hormone gene. Mol Cell Endocrinol 1999 ; 154 : 137-49.
- 37. Harris M, Aschkenasi C, Elias CF, et al. Transcriptional regulation of the thyrotropin-releasing hormone gene by leptin and melanocortin signaling. J Clin Invest 2001 ; 107 : 111-20.
- 38. Sasaki S, Lesoon-Wood LA, Dey A, et al. Ligand-induced recruitment of a histone deacetylase in the negative-feedback regulation of the thyrotropin beta gene. EMBO J 1999 ; 18 : 5389-98.
List of figures
Figure 1
Mécanisme de rétrocontrôle négatif de la synthèse des hormones thyroïdiennes.
Les hormones thyroïdiennes T3 et T4 synthétisées par la thyroïde sont transportées dans le sang par des protéines plasmatiques. La T4 étant la forme majoritaire produite par la thyroïde, des désiodases vont contribuer à la désiodation de la T4 en T3, forme biologiquement active. Cette dernière agit dans les tissus périphériques en contrôlant la transcription de ses gènes cibles ; de plus, elle contrôle négativement l’expression des gènes codant pour la TSH (thyréostimuline) et la TRH (thyréolibérine), stimulatrices de la sécrétion des hormones thyroïdiennes par la thyroïde.
Figure 2
Activation transcriptionnelle par des récepteurs d’hormones thyroïdiennes fixés sur des TRE positifs (TREp).
A. En l’absence de ligand (T3), les TR (appelés dans ce cas apo-TR) hétérodimérisés avec le facteur RXR recrutent un complexe multiprotéique corépresseur comprenant notamment des histone désacétylases (HDAC), responsables du maintien de la chromatine dans un état condensé peu favorable à la transcription. D’autres protéines interagissent également avec les cofacteurs de la machinerie transcriptionnelle de base, entraînant ainsi une répression de la transcription du gène cible. B. La liaison de la T3 aux TR (qualifiés dans ce cas d’holo-TR) induit leur changement conformationnel, permettant un relargage du complexe corépresseur au bénéfice de complexes coactivateurs comprenant des enzymes de modification des histones de type HAT (CBP/p300) et des protéines TRAP/DRIP probablement associées à d’autres coactivateurs interagissant avec la machinerie transcriptionnelle (ARNpol II + TAF + TFIIB +TBP), l’ensemble permettant une activation de la transcription (voir texte). CBP : protéine de liaison à CREB ; DRIP : protéine interagissant avec le récepteur de la vitamine D ; GTF : facteur général de transcription ; HDAC : histone désacétylase ; HAT : histone acétyltransférase ; p/CAF : facteur associé à p300/CBP ; RXR : récepteur de l’acide 9-cis-rétinoïque ; NcoR : corépresseur nucléaire ; SRC : coactivateur des récepteurs stéroïdiens ; TAF : facteur associé à TBP ; TBP : protéine de liaison à la boîte TATA ; TF : facteur de transcription ; TR : récepteur des HT ; TRAP : protéine associée aux TE ; TREp : élément de réponse positif aux TR (d’après [11]).
Figure 3
Modèle hypothétique de répression transcriptionnelle du gène TRH par des TRβ liés aux TRE négatifs (TREn).
A. En l’absence de T3, le gène TRH est activé par une ou plusieurs voies faisant notamment intervenir la protéine CREB et les cofacteurs qui lui sont associés. L’encombrement stérique créé par CREB et ses coactivateurs au niveau du site CRE empêcherait les TRβ de se fixer à leur TREn. Les TR, capables de recruter des complexes corépresseurs en l’absence de T3, séquestreraient ces derniers soit « en solution », soit au niveau de régions régulatrices distales du site d’initiation de la transcription. TRβ1 possède également la capacité d’induire une activation du gène TRH indépendante de T3, ce qui impliquerait peut-être la participation des facteurs cités ci-dessus, protéine CREB et coactivateurs associés [25]. B. En présence de T3, les isoformes TRβ seraient capables de se lier au TREn du gène TRH. La répression pourrait être induite via le recrutement d’une ou plusieurs protéines, peut-être spécifiques de l’hypothalamus, capables de réprimer la transcription. La possibilité que TRβ1 interagisse avec HDAC2 en présence de ligand est envisageable, cette dernière pouvant désacétyler les histones et contribuer ainsi à la répression transcriptionnelle [38]. Cela vient d’ailleurs d’être démontré dans une lignée cellulaire (qui n’exprime pas, normalement, de TRH), dans laquelle HDAC2 serait recrutée en présence de T3, provoquant la désacétylation transitoire des histones et un relargage de NCoR initialement associé à TR au niveau du promoteur TRH [31]. CRE : élément de réponse à l’AMPc ; CREB : protéine de liaison à CRE ; HDAC : histone désacétylase ; CBP/p300 : protéine de liaison à CREB ; RXR : récepteur de l’acide 9-cis-rétinoïque ; CoR : corépresseur; TR : récepteur des HT ; TREn : élément de réponse négatif aux TR.