Article body
L’épissage des gènes est un passage quasi obligé de l’expression génique chez les eucaryotes supérieurs. De plus, pour les trois quarts des gènes de mammifères [1], il existe des possibilités alternatives, parfois nombreuses, d’épissage de chaque transcrit primaire, ce qui rend compte au moins d’une grande partie de l’écart entre le nombre de gènes (environ 30 000) et celui des protéines qui est environ dix fois supérieur [2]. L’épissage alternatif est donc un niveau essentiel de la régulation qualitative de l’expression des gènes puisqu’il permet à une même séquence d’ADN de produire des protéines différentes selon le tissu ou les conditions d’environnement. Mais, en contrepartie de son rôle créateur de diversité protéique physiologique, l’épissage alternatif est aussi une occasion de produire des transcrits aberrants aux conséquences pathologiques très variées.
Épissage alternatif et dégénérescence des sites d’épissage
Pour faire face à la multiplicité des sites potentiels d’épissage et à leur faible conservation, la machinerie d’épissage, le spliceosome, doit être doué d’une grande flexibilité (pour une revue récente, voir [3]) et comporter à la fois des éléments communs à tous les sites d’épissage et d’autres, spécifiques de certains d’entre eux, la combinatoire de ces différents éléments réalisant un véritable « code de l’épissage » permettant un choix régulé des épissages alternatifs. Outre les jonctions intron-exon qui sont insuffisantes pour définir les sites d’épissage, ceux-ci requièrent des éléments supplémentaires de deux types : (1) des éléments cis qui sont des séquences régulatrices activatrices ou inhibitrices et peuvent être localisées dans les exons aussi bien que dans les introns, d’où quatre catégories : ESE et ESS d’une part (exonic splicing enhancer et exonic splicing silencer), ISE et ISS (intronic splicing enhancer et intronic splicing silencer), d'autre part ; (2) des facteurs trans capables de reconnaître ces séquences et qui seront donc soit des répresseurs, soit des activateurs.
La dégénérescence des sites d’épissage, indispensable à l’épissage alternatif, ne va pas sans inconvénients. Elle a en effet pour conséquence l’existence de nombreux sites cryptiques (en particulier dans les introns qui sont très longs) normalement trop faibles pour être utilisés de façon significative, mais qui peuvent facilement accéder à la compétence à la faveur de simples mutations ponctuelles. À l’inverse, ces mêmes mutations peuvent tout aussi bien inactiver des sites physiologiques. On observe d’ailleurs de plus en plus d’épissages aberrants à l’origine de maladies génétiques. Dans cette optique, il importe d’avoir présent à l’esprit que certaines de ces mutations, jusqu’alors considérées comme de simples polymorphismes car n’affectant pas la séquence codante, peuvent avoir en fait un retentissement profond donc pathologique sur la structure des protéines par le biais d’altérations de l’épissage.
Les facteurs protéiques répresseurs incluent des protéines liées aux hnRNP (heterogenous ribonucleoprotein), tandis que les facteurs stimulateurs appartiennent à la famille des protéines SR. Celles-ci sont caractérisées par un domaine de reconnaissance de l’ARN (RRM, RNA recognition motif) et un domaine RS ainsi appelé pour sa richesse en résidus arginine et sérine et qui est engagé à la fois dans des interactions protéine-protéine, et aussi avec l’ARN. L’abondance des résidus sérine confère aux protéines SR une grande capacité de phosphorylation qui affecte la régulation de l’épissage alternatif [4].
Les protéines SR comme nouvelle cible thérapeutique
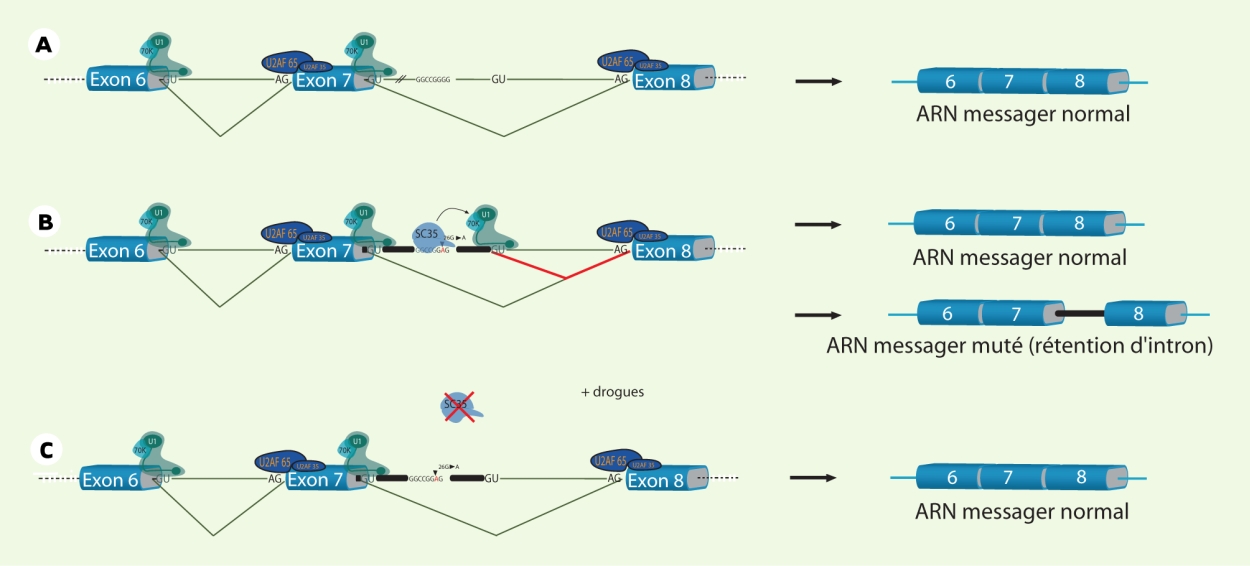
Les protéines SR ont donc une double spécificité qui leur permet : (1) de contribuer à la définition des exons et à la sélection des sites d’épissage par la reconnaissance des séquences régulatrices de l’ARN pré-messager ; (2) d’y recruter la machinerie d’épissage (spliceosome) par des interactions protéine-protéine. Ainsi, la création par mutation d’un nouvel ESE pour une protéine SR donnée aboutira à l’inclusion de séquences normalement introniques avec des conséquences pathologiques évidentes. C’est le cas du syndrome de Leigh (encéphalomyélopathie avec retard mental et acidose lactique) où une mutation dans l’intron 7 de la pyruvate déshydrogénase E1α crée un site de forte affinité pour la protéine SR SC35 activant à son voisinage un site d’épissage normalement cryptique, avec pour conséquence l’inclusion de séquences introniques inactivant l’enzyme (Figure 1).
Figure 1
Correction d’une altération d’épissage responsable du syndrome de Leigh.
Survenant dans la première année de l’enfant, le syndrome de Leigh provoque des troubles neurologiques et moteurs très sévères. À l’origine de cette maladie, une carence dans la production d’énergie des cellules de l’organisme. Plusieurs gènes ont été mis cause dont des gènes codant pour les sous-unités de la pyruvate déshydrogénase (PDH). Le cas présenté sur ce schéma concerne une nouvelle mutation dans l’intron 7 du gène PDH E1α chez un patient atteint d’encéphalopathie et d’acidose lactique. A. Représentation schématique des exon 6, 7 et 8 (rectangle bleu) et des introns 6 et 7 (trait). La particule U1 snRNP et les deux sous-unités du facteur U2AF (U2AF 65 et U2AF 35) sont fixées respectivement aux sites d’épissage 5’ et 3’ pour permettre la reconnaissance de ces sites par la machinerie d’épissage et ainsi favoriser l’assemblage du spliceosome autour de ces sites. Les dinucléotides GU et AG, caractéristiques de la plupart des terminaisons introniques, sont également représentés. L’épissage physiologique qui utilise ces sites canoniques donne lieu à un seul messager. B. Chez le patient, outre l’ARN PDH E1α correctement épissé, il existe un messager aberrant qui résulte de l’utilisation d’un site donneur d’épissage cryptique localisé en aval de la mutation. En conséquence, cet ARN contient 45 nucléotides de l’intron 7 insérés en phase entre les exons 7 et 8. La mutation intronique située 26 nucléotides en aval de l’exon 7 transforme un G en A. Ce changement crée un site de haute affinité pour la fixation de la protéine SR SC35 (séquence mutée GGCCGGAG au lieu de la séquence sauvage GGCCGGAG) et ainsi permet à SC35 de recruter la snRNP U1 sur un site cryptique situé 19 nucléotide en aval de la mutation. Deux messagers sont donc produits : l’un correspondant au message sauvage, l’autre portant une insertion de 45 nucléotides de l’intron 7. C. L’utilisation de petites molécules qui interfèrent avec l’activité de SC35 a permis in vitro de supprimer l’épissage aberrant tout en maintenant l’épissage physiologique.
Afin de contribuer à l’identification de nouveaux médicaments capables d’interférer avec des épissages pathologiques, nous avons entrepris de rechercher des petites molécules chimiques ciblant le processus d’épissage. Notre premier fil conducteur a été de rechercher des inhibiteurs de l’activité de phosphorylation des protéines SR que nous venions de découvrir associée à l’ADN topo-isomérase I [5]. Certaines, parmi les molécules actives, touchaient directement cette enzyme [6], d’autres, les protéines SR qui en sont les substrats [7]. Ces dernières étaient particulièrement intéressantes car elles ouvraient la possibilité de trouver des molécules spécifiques de différentes protéines SR et donc de différentes catégories d’épissage alternatif. Plusieurs molécules inhibant spécifiquement les épissages dépendant de plusieurs de ces protéines (ASF/SF2, SC35 et SRp55), et appartenant toutes à la famille des dérivés de l’indole, ont pu être déjà sélectionnées.
Comme première étape de validation de ces molécules pour supprimer des épissages aberrants, nous avons utilisé les cellules d’un malade atteint du syndrome de Leigh résultant d’une inactivation de la pyruvate déshydrogénase E1α (Figure 1). Après avoir précédemment montré qu’il était possible de supprimer la production de la protéine anormale en réduisant artificiellement par un siARN la quantité de protéine SC35 dans les cellules [8], notre dernier travail établit maintenant que deux molécules dérivées de l’indole, et sélectionnées pour inhiber spécifiquement la protéine SC35, bloquent effectivement l’utilisation du site cryptique à l’origine de la protéine pathologique et restaurent celle du site normal [7].
L’intérêt des maladies génétiques comme cible d’utilisation de ces petites molécules est de se prêter à des « preuves de concept » dans des situations parfaitement définies où un événement unique crée la pathologie. Un autre champ d’application thérapeutique pourrait être les infections par des virus dont la production implique des épissages alternatifs multiples (sida, adénovirus…). Nous montrons en effet que ces molécules bloquent la production des ARN viraux du VIH-1 dans des cellules chroniquement infectées. Quant au cancer, qui est susceptible de représenter une cible particulièrement importante en raison de la multiplicité des événements d’épissage alternatif, celle-ci même rend la situation très complexe et impose un travail important de défrichage préalable.
Appendices
Références
- 1. Johnson JM, Castle J, Garrett-Engele P, et al. Genome-wide survey of human alternative pre-mRNA splicing with exon junction microarrays. Science 2003 ; 302 : 2141-4.
- 2. International Human Genome Sequencing Consortium. Finishing the euchromatic sequence of the human genome. Nature 2004 ; 431 : 931-45.
- 3. Tazi J, Durand S, Jeanteur P. Spliceosome: a novel multi-faceted target for therapy. Trends Biochem Sci 2005 ; 30 : 469-78.
- 4. Soret J, Tazi, J. Phosphorylation-dependent control of the pre-mRNA splicing machinery. Prog Mol Subcell Biol 2003 ; 31 : 89-126.
- 5. Rossi F, Labourier E, Forne T, et al. Mammalian DNA topoisomerase I specifically phosphorylates SR protein splicing factors. Nature 1996 ; 381 : 80-2.
- 6. Tazi J, Bakkour N, Soret J, et al. Selective inhibition of topoisomerase I and various steps of spliceosome assembly by diospyrin derivatives. Mol Pharmacol 2005 ; 67 : 1186-94.
- 7. Soret J, Bakkour N, Maire S, et al. Selective modification of alternative splicing by indole derivatives that target SR protein splicing factors. Proc Natl Acad Sci USA 2005 ; 102 : 8764-9.
- 8. Gabut M, Mine M, Marsac C, et al. The SR protein SC35 is responsible for aberrant splicing of the E1α pyruvate dehydrogenase mRNA in a case of mental retardation with lactic acidosis. Mol Cell Biol 2005 ; 8 : 3286-94.
List of figures
Figure 1
Correction d’une altération d’épissage responsable du syndrome de Leigh.
Survenant dans la première année de l’enfant, le syndrome de Leigh provoque des troubles neurologiques et moteurs très sévères. À l’origine de cette maladie, une carence dans la production d’énergie des cellules de l’organisme. Plusieurs gènes ont été mis cause dont des gènes codant pour les sous-unités de la pyruvate déshydrogénase (PDH). Le cas présenté sur ce schéma concerne une nouvelle mutation dans l’intron 7 du gène PDH E1α chez un patient atteint d’encéphalopathie et d’acidose lactique. A. Représentation schématique des exon 6, 7 et 8 (rectangle bleu) et des introns 6 et 7 (trait). La particule U1 snRNP et les deux sous-unités du facteur U2AF (U2AF 65 et U2AF 35) sont fixées respectivement aux sites d’épissage 5’ et 3’ pour permettre la reconnaissance de ces sites par la machinerie d’épissage et ainsi favoriser l’assemblage du spliceosome autour de ces sites. Les dinucléotides GU et AG, caractéristiques de la plupart des terminaisons introniques, sont également représentés. L’épissage physiologique qui utilise ces sites canoniques donne lieu à un seul messager. B. Chez le patient, outre l’ARN PDH E1α correctement épissé, il existe un messager aberrant qui résulte de l’utilisation d’un site donneur d’épissage cryptique localisé en aval de la mutation. En conséquence, cet ARN contient 45 nucléotides de l’intron 7 insérés en phase entre les exons 7 et 8. La mutation intronique située 26 nucléotides en aval de l’exon 7 transforme un G en A. Ce changement crée un site de haute affinité pour la fixation de la protéine SR SC35 (séquence mutée GGCCGGAG au lieu de la séquence sauvage GGCCGGAG) et ainsi permet à SC35 de recruter la snRNP U1 sur un site cryptique situé 19 nucléotide en aval de la mutation. Deux messagers sont donc produits : l’un correspondant au message sauvage, l’autre portant une insertion de 45 nucléotides de l’intron 7. C. L’utilisation de petites molécules qui interfèrent avec l’activité de SC35 a permis in vitro de supprimer l’épissage aberrant tout en maintenant l’épissage physiologique.