Article body
Il est admis qu'après la transcription de l'ADN en ARNm, plusieurs protéines se fixent sur des séquences/structures particulières de l'ARNm. Ces complexes dictent le destin de chaque ARNm quant à sa stabilité, son transport et sa traduction [1]. Staufen1 est l'une de ces protéines qui lient des structures double-brin de l'ARNm [2-4]. De nombreux travaux tant chez les mammifères que chez le xénope ou la drosophile ont montré le rôle joué par Staufen1 dans le transport et la localisation des ARNm dans les ovocytes et les neurones. Ces phénomènes de localisation de l'ARN et de traduction locale sont essentiels, entre autres, pour l'établissement des axes embryonnaires, la division asymétrique des neuroblastes et la plasticité synaptique [6].
Chez les mammifères, Staufen1 se retrouve majoritairement associé aux ribosomes et/ou au réticulum endoplasmique rugueux [3, 4]. Dans les neurones, il est retrouvé dans les granules d'ARN du corps cellulaire et des dendrites [7]. Récemment, nous avons isolé des complexes ribonucléoprotéiques contenant Staufen1 et identifié plusieurs ARNm associés à ces complexes [8]. Parmi ceux-ci, l'ARNm du facteur de ribosylation de l'ADP (Arf1) codant pour une GTPase impliquée dans le transport vésiculaire a été étudié plus en détail (voir plus loin).
Staufen1 interagit avec Upf1
Parallèlement, nous avons récemment identifié Staufen1 dans un criblage visant la recherche de partenaires protéiques interagissant avec Upf1 [8], un facteur impliqué dans la dégradation des ARNm porteurs de codon non-sens (NMD, non-sense mediated RNA decay). Le NMD est un mécanisme de contrôle de la qualité des ARNm [9]. Lors de la transcription, des erreurs dans l'incorporation des nucléotides peuvent engendrer des mutations donnant lieu à l'apparition de codon non-sens à l'intérieur de la séquence codante (Figure 1A). La traduction de ces ARNm mutés créerait des protéines tronquées ayant un potentiel néfaste pour la cellule.
Figure 1
Comparaison des mécanismes NMD et SMD.
A. Lors de l'épissage des transcrits primaires, un complexe protéique (EJC, exon junction complex) se dépose une vingtaine de nucléotides en amont des jonctions exon-exon (JEE). Si ce complexe composé de plusieurs protéines dont les facteurs Upf3X et Upf2 recrute le facteur Upf1, le NMD sera déclenché. B. (1) Les transcrits porteurs de codons non-sens apparus à la suite d'une erreur de transcription sont éliminés par le NMD. Staufen1 n'est pas impliqué dans ce mécanisme. (2-3) Le recrutement de Staufen1 au moyen d'une protéine de fusion Staufen1-MS2 (MS2 étant une protéine qui lie une structure spécifique de l'ARN) sur la séquence de reconnaissance MS2 (MBS, MS2-binding site) déclenche le SMD si celle-ci est placée en aval du codon normal de terminaison. (4) Staufen1, en se liant à une séquence d'ARN (SBS, Staufen1-binding site) située en aval du codon de terminaison normal sur le transcrit endogène Arf1 déclenche le SMD. (5) Cette séquence est importante puisque sa délétion protège le transcrit Arf1 de la dégradation. (6) De même, un transcrit rapporteur sur lequel a été fusionnée la séquence SBS devient sensible au SMD.
Pour déterminer si Staufen1 faisait partie du mécanisme NMD, nous avons étudié l'effet de la sous-expression de Staufen1 sur l'abondance d'un ARNm qui contient un codon non-sens dans sa région codante et qui est par conséquent sensible à la dégradation par le NMD (Figure 1B-1). La sous-expression, par la technique des ARN interférents [10], d'un facteur important pour le NMD devrait stabiliser ce transcrit et en augmenter la quantité dans la cellule. Nous avons constaté que la sous-expression de Staufen1 ne change pas le niveau de ce transcrit, contrairement à ce qui se produit après la sous-expression de l'un ou l'autre des différents facteurs Upf impliqués dans le NMD. Ces résultats ne plaident pas en faveur d'un rôle joué par Staufen1 dans le NMD.
Staufen1 induit la dégradation des ARNm normaux auxquels il est lié
Si Staufen1 n'a pas d'effet sur le NMD, quelle est la signification de son association avec Upf1? Pour répondre à cette question, nous avons d'abord montré que la sous-expression de Staufen1 résulte en une augmentation du niveau stationnaire du transcrit endogène Arf1 [8]. Cette augmentation ne vient pas d'un effet transcriptionnel, mais de l'allongement de la demi-vie du transcrit. Ce résultat montre que Staufen1 est impliqué dans une voie qui module la stabilité/dégradation des ARN auxquels il est lié.
Par quel mécanisme Staufen1 peut-il moduler la stabilité/dégradation des transcrits auxquels il est lié ? Serait-il possible que la simple liaison de Staufen1 en aval d'un codon de terminaison normal soit suffisante pour provoquer la dégradation du transcrit ? Nous avons testé cette hypothèse par différentes approches [8]. Tout d'abord, nous avons recruté Staufen1 sur un transcrit rapporteur et avons observé la dégradation du transcrit uniquement lorsque Staufen1 se lie en aval du codon de terminaison normal (Figure 1B-2, 1B-3). De même, nous avons montré que la séquence d'ARN reconnue par Staufen1 est située dans la région 3' non traduite, en aval du codon de terminaison normal (Figure 1B-4). Cette séquence est requise pour la dégradation de l'ARNm puisque le transcrit Arf1 n'est plus dégradé après délétion de cette séquence (Figure 1B-5). De même, sa fusion dans la région 3' non traduite d'un transcrit rapporteur est suffisante pour induire la dégradation de celui-ci (Figure 1B-6). La déstabilisation observée dans ces expériences est annulée à la suite de la sous-expression du facteur Upf1 mais non consécutivement à celle des facteurs Upf2 ou Upf3X. Ce résultat est en accord avec le fait que Staufen1 interagit directement avec Upf1 mais non avec les autres facteurs du NMD et que Upf1 agit en aval des autres facteurs devant intervenir dans la cascade qui mène à la dégradation de l'ARNm.
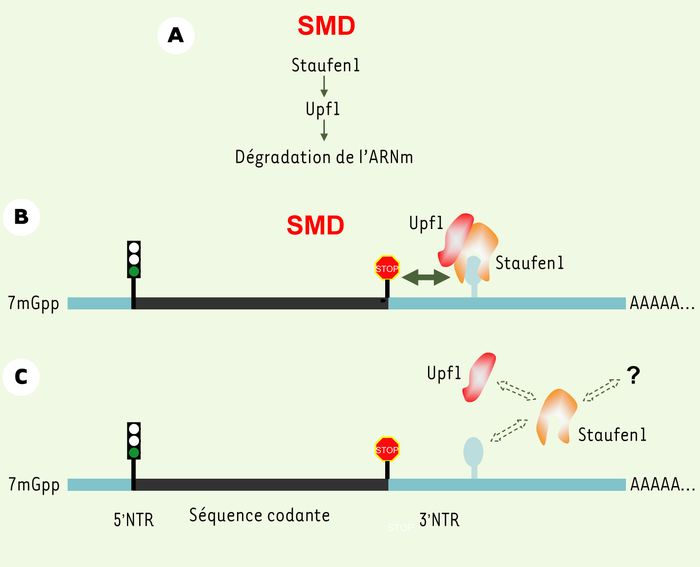
Tous ces résultats nous permettent de proposer un nouveau mécanisme que l'on nomme SMD (Staufen1-mediated mRNA decay). La sous-population d'ARNm qui porte un site de reconnaissance pour Staufen1 peut recruter le complexe Staufen1-Upf1 (Figure 2A). Si ce complexe se lie à l'ARNm en aval du codon de terminaison normal, la dégradation de l'ARNm sera déclenchée (Figure 2B). La présence ou non sur un transcrit de la séquence de liaison à Staufen1 et sa position par rapport à celle du codon de terminaison normal dictent le déclenchement ou non du SMD.
Figure 2
Modèle de fonctionnement du SMD.
A. La liaison du complexe Staufen1-Upf1 sur un transcrit peut induire la dégradation de ce transcrit. Le SMD ne requiert pas de jonction exon-exon, ni la présence des facteurs du complexe EJC. B. Lorsque le complexe Staufen1/Upf1 se lie en aval d'un codon de terminaison normal au niveau de la région 3'-non traduite (NTR) d'un ARNm, il entraîne la dégradation de celui-ci (flèche noire). C. Le SMD pourrait être modulé au niveau de l'association entre Staufen1 et Upf1, de la liaison de Staufen1 à l'ARNm et/ou de la modification post-traductionnelle de Staufen1 par des facteurs encore inconnus.
Rôle du SMD
Nos résultats montrent que le SMD est un nouveau mécanisme de contrôle post-transcriptionnel de l'expression des gènes normaux et non un mécanisme d'élimination de codons non-sens apparus à la suite d'une erreur de transcription [11]. Le SMD pourrait réguler l'expression d'une sous-population de transcrits définie par sa capacité à lier Staufen1. Le choix entre le transport, la dégradation ou la traduction de ces messagers pourrait être régi par les besoins de la cellule, en modulant soit l'association entre Staufen1 et Upf1, soit l'association entre Staufen1 et cette sous-population d'ARNm (Figure 2C). Il sera donc intéressant de déterminer l'importance du SMD pour la physiologie cellulaire et de mieux comprendre comment la cellule utilise Staufen1 autant pour la localisation de l'ARNm que pour sa dégradation.
Appendices
Remerciements
Ce projet de recherche résulte d'une collaboration avec une autre équipe impliquant Yoon Ki Kim et Lynne E. Maquat (Department of Biochemistry and Biophysics, School of Medicine and Dentistry, University of Rochester, 601 Elmwood Avenue, Box 712, Rochester, NY 14642, États-Unis).
Références
- 1. Siomi H, Dreyfuss G. RNA-binding proteins as regulators of gene expression. Curr Opin Genet Dev 1997 ; 7 : 345-53.
- 2. St Johnston D, Beuchle D, Nüsslein-Volhard C. Staufen, a gene required to localize maternal RNAs in the Drosophila egg. Cell 1991 ; 66 : 51-63.
- 3. Wickham L, Duchaîne T, Luo M, et al. Mammalian staufen is a double-stranded RNA- and tubulin-binding protein which localizes to the rough endoplasmic reticulum. Mol Cell Biol 1999 ; 19 : 2220-30.
- 4. Marion RM, Fortes P, Beloso A, et al. A human sequence homologue of Staufen is an RNA-binding protein that is associated with polysomes and localizes to the rough endoplasmic reticulum. Mol Cell Biol 1999 ; 19 : 2212-9.
- 5. DesGroseillers L, Duchaîne T, Luo M. Transport et localisation d'ARN messagers chez les mammifères : rôle de la protéine staufen. Med Sci(Paris) 1999 ; 15 : 1164-7.
- 6. Kiebler MA, DesGroseillers L. Molecular insights into mRNA transport and local translation in the mammalian nervous system. Neuron 2000 ; 25 : 19-28.
- 7. Köhrmann M, Luo M, Kaether K, et al. Microtubule-dependent recruitment of Staufen-GFP into large granules and subsequent dendritic transport in living hippocampal neurons. Mol Biol Cell 1999 ; 10 : 2945-53.
- 8. Kim YK, Furic L, DesGroseillers L, Maquat LE. Mammalian Staufen1 recruits Upf1 to specific mRNA 3'UTRs so as to elicit mRNA decay. Cell 2005 ; 120 : 195-208.
- 9. Maquat LE. Nonsense-mediated mRNA decay: splicing, translation and mRNP dynamics. Nat Rev Mol Cell Biol 2004 ; 5 : 89-99.
- 10. Hannon GJ, Rossi JJ. Unlocking the potential of the human genome with RNA interference. Nature 2004 ; 431 : 371-8.
- 11. Meyer EL, Gavis ER. Staufen does double duty. Nat Struct Mol Biol 2005 ; 12 : 291-2.
List of figures
Figure 1
Comparaison des mécanismes NMD et SMD.
A. Lors de l'épissage des transcrits primaires, un complexe protéique (EJC, exon junction complex) se dépose une vingtaine de nucléotides en amont des jonctions exon-exon (JEE). Si ce complexe composé de plusieurs protéines dont les facteurs Upf3X et Upf2 recrute le facteur Upf1, le NMD sera déclenché. B. (1) Les transcrits porteurs de codons non-sens apparus à la suite d'une erreur de transcription sont éliminés par le NMD. Staufen1 n'est pas impliqué dans ce mécanisme. (2-3) Le recrutement de Staufen1 au moyen d'une protéine de fusion Staufen1-MS2 (MS2 étant une protéine qui lie une structure spécifique de l'ARN) sur la séquence de reconnaissance MS2 (MBS, MS2-binding site) déclenche le SMD si celle-ci est placée en aval du codon normal de terminaison. (4) Staufen1, en se liant à une séquence d'ARN (SBS, Staufen1-binding site) située en aval du codon de terminaison normal sur le transcrit endogène Arf1 déclenche le SMD. (5) Cette séquence est importante puisque sa délétion protège le transcrit Arf1 de la dégradation. (6) De même, un transcrit rapporteur sur lequel a été fusionnée la séquence SBS devient sensible au SMD.
Figure 2
Modèle de fonctionnement du SMD.
A. La liaison du complexe Staufen1-Upf1 sur un transcrit peut induire la dégradation de ce transcrit. Le SMD ne requiert pas de jonction exon-exon, ni la présence des facteurs du complexe EJC. B. Lorsque le complexe Staufen1/Upf1 se lie en aval d'un codon de terminaison normal au niveau de la région 3'-non traduite (NTR) d'un ARNm, il entraîne la dégradation de celui-ci (flèche noire). C. Le SMD pourrait être modulé au niveau de l'association entre Staufen1 et Upf1, de la liaison de Staufen1 à l'ARNm et/ou de la modification post-traductionnelle de Staufen1 par des facteurs encore inconnus.