Abstracts
Résumé
Le facteur de transcription TBP (TATA-binding protein) est une sous-unité de plusieurs complexes macromoléculaires nécessaires à la transcription par les trois ARN polymérases nucléaires. Cette observation avait conduit à faire de TBP un facteur « universel » de la transcription. La dé-couverte de trois protéines apparentées à TBP, et d’un complexe macromoléculaire dépourvu de TBP, mais capable d’initier la transcription par l’ARN polymérase II in vitro, a conduit à remettre en cause le caractère universel de TBP. Plusieurs études in vivo ont montré que TBP a plutôt un rôle specifique dans l’activation de certains gènes impliqués dans la prolifération cellulaire. De plus, le domaine aminoterminal de TBP joue un rôle dans l’établissement de la tolérance foetomaternelle.
Summary
The TATA binding protein (TBP) is a subunit of several macromolecular complexes required for transcription by the three nuclear RNA polymerases. This observation led to the idea that TBP is a « universal » transcription factor. The discovery of three TBP-related factors and a macromolecular complex which lacks TBP but can support RNA polymerase II transcription in vitro has led to a reappraisal of the universal character of TBP. Several in vivo studies have rather shown that TBP plays a specific role in the activation of a subset of cellular genes controlling the cell cycle. In mammals, the aminoterminal region of TBP plays a highly selective role in the maternal immunotolerance of pregnancy.
Article body
L’initiation de la transcription par les ARN polymérases fait intervenir une machinerie complexe, mais conservée au cours de l’évolution. La protéine TBP (TATA binding protein) est un des facteurs clés dans ce processus. En effet, TBP paraît essentiel pour la transcription par les trois ARN polymérases, pol I, pol II et pol III [1-5].
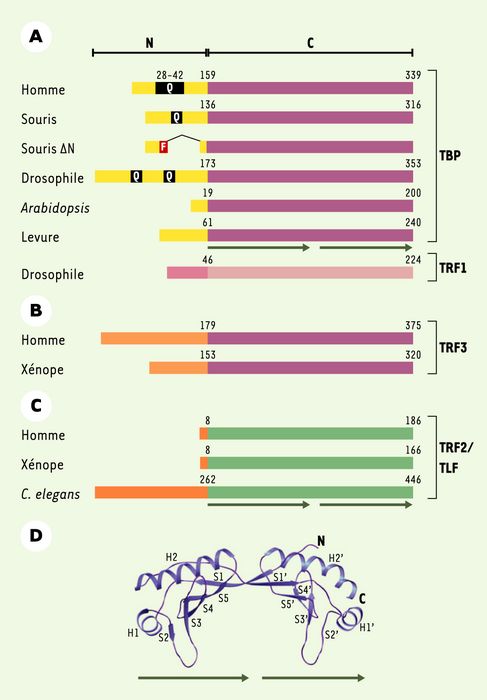
TBP reconnaît l’élément TATA en amont du site d’initiation d’un grand nombre de promoteurs (Figure 1). C’est une protéine bipartite, avec une région aminoterminale variable et un domaine carboxyterminal très conservé. Le domaine aminoterminal présente une importante variabilité de séquence et de taille en fonction des espèces (Figure 2A). Le domaine carboxyterminal est conservé à près de 80 % entre la levure et l’homme. Il adopte une structure en « selle de cheval » (Figure 2D) avec une face concave hydrophobe interagissant avec l’ADN et une face convexe qui interagit avec TFIIA, TFIIB et autres facteurs [6-10].
Figure 1
Structure des promoteurs des gènes transcrits par les ARN polymérases I, II et III.
TBP (TATA binding protein) est une sous-unité des complexes nécessaires à la transcription par les trois ARN polymérases. A. La transcription par l’ARN polymérase I (Pol I) des gènes codant pour les ARN ribosomiques (ARNr) nécessite la fixation de UBF (upstream binding factor) à l’élément régulateur UE (upstream element) et la fixation du complexe SL1 (selectivity factor 1), contenant TBP et trois TAF (TBP-associated factors), au promoteur proximal CE (core element). B. La transcription par l’ARN polymérase II (pol II) des ARNm peut faire intervenir le complexe TFIID (transcription factor IID), formé de TBP et de treize TAF (représentés ici par un seul disque). C. La transcription par pol II peut aussi se faire par l’intermédiaire du complexe B-TFIID formé de TBP et BTAF1. D. TRF3 (TBP-related factor) est un nouveau facteur impliqué dans la transcription par pol II. Son interaction avec les TAF reste à démontrer. E. La transcription par pol III fait intervenir les complexes TFIIIA et TFIIIC qui se fixent sur les éléments intragéniques, par exemple du gène codant pour les ARN ribosomiques 5S (ARN 5S) comme indiqué ici. Le complexe TFIIIB contenant TBP se fixe en amont du gène. F. Dans le cas de la transcription du gène codant pour les petits ARN nucléaires U6 (ARN U6), TBP se fixe sur l’élément TATA et interagit avec le complexe SNAPc (small nuclear RNA-activating protein complex) fixé sur PSE (proximal sequence element) par l’intermédiaire de son domaine aminoterminal. L’activation du gène ARN U6 se fait par le facteur Oct1 (octamer-binding transcription factor 1) fixé à une séquence OCTA (octamer). Pour plus de clarté, les cinq sous-unités du complexe SNAPc sont réprésentées par un seul disque. BRF2 : TFIIB-related factor, subunit of RNA polymerase III transcription initiation factor.
Figure 2
Organisation et structure de la famille des TBP (TATA binding proteins).
A. Organisation de TBP de différentes espèces. Le domaine aminoterminal est réprésenté en jaune et la présence des répétitions des résidus glutamines (Q) en noir. Le polymorphisme allélique humain (28-42 glutamines) est indiqué. La structure du mutant TBPΔN murin (souris ³N) mentionné dans l’article ainsi que l’épitope FLAG (étiquette génétique) (F) sont également schématisés. L’alignement de TRF1 (TBP-related factor 1) et de la famille TBP sont présentés. Le premier acide aminé du domaine conservé est indiqué pour chacun des TBP. B, C. Alignement de TRF3 et TRF2/TLF (TBP-like factor) et de la famille TBP. La répétition directe qui forme la structure en selle de cheval du domaine conservé est indiquée par les flèches. D. Représentation de la structure en selle de cheval du domaine carboxyterminal conservé de TBP, déterminée par cristallographie aux rayons X. H1, H1’, H2 et H2’ représentent les hélices α et S1-S5 et S1’-S5’ les feuillets β.
Sa forte conservation et sa présence dans les complexes d’initiation des trois ARN polymérases ont fait de TBP le « facteur de transcription universel ». Des découvertes récentes ont changé notre perspective sur ce caractère universel de TBP.
Des protéines apparentées à TBP
La remise en cause du caractère universel de TBP est venue de la découverte de protéines apparentées à TBP. En effet, trois protéines TRF1 (TBP-related factor 1), TLF (TBP-like factor)/TRF2 et TRF3 ont été identifiées et caractérisées [11-17]. La similarité entre ces protéines est limitée au domaine carboxyterminal conservé (Figure 2A-C). TRF2 présente 60 % de similarité et 40 % d’identité avec TBP, mais TRF1 et TRF3 sont encore plus proches de TBP. Dans tous ces facteurs, les sites d’interaction de TFIIA et de TFIIB sont conservés et TRF1 et TRF3 se fixent sur les séquences TATA. En revanche, TRF2 ne fixe pas les éléments TATA [11] et aucun site de fixation de TRF2 n’est actuellement connu.
TRF1
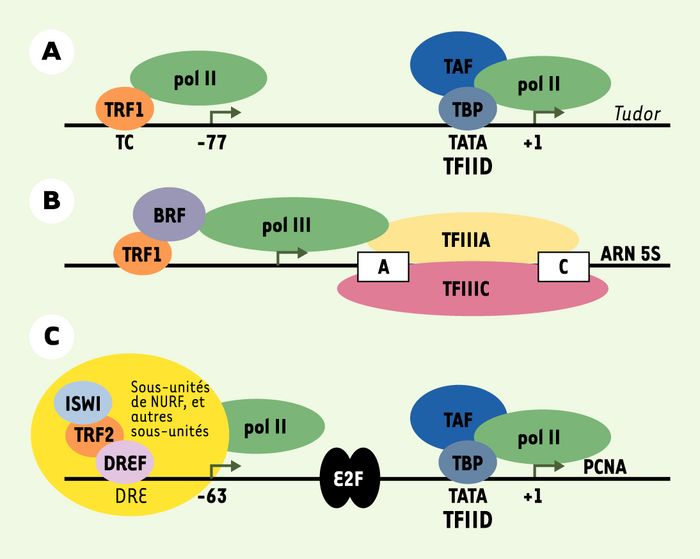
TRF1 n’existe que chez la drosophile où il est exprimé pendant l’embryogenèse puis, de façon spécifique, dans le système nerveux central et les cellules germinales chez l’adulte [16, 18]. TRF1 agit comme un facteur de transcription spécifique pour l’expression d’un sous-ensemble de gènes dont Tudor [18]. TRF1 dirige la transcription de Tudor à partir d’un deuxième site d’initiation en amont de celui dirigé par TFIID (Figure 3A). De plus, TRF1 interagit avec BRF (TFIIB-related factor), un facteur de transcription pol III, et semble se substituer à TBP pour la transcription pol III dans cet organisme [19].
Figure 3
Transcription par TRF1 et TRF2 (TBP-related factors 1 et 2).
A. La structure du promoteur du gène Tudor est schématisée ainsi que les complexes qui interviennent à chaque site d’initiation. B. Schéma du gène codant pour les ARN ribosomiques 5S (ARN 5S) (voir Figure 1E) sous le contrôle du complexe TRF1-BRF (TFIIB-related factor). C. La structure du promoteur du gène PCNA(proliferating cell nuclear antigen) est schématisée montrant la présence des sites de fixation TATA, E2F (facteur de transcription E2F) et DRE (DNA replication-related element). Le complexe TFIID (transcription factor IID) initie la transcription au promoteur proximal et le complexe TRF2 au promoteur distal, une organisation analogue à la transcription de Tudor à partir de deux sites d’initiation par TFIID et TRF1. TAF : TBP-associated factor ; NURF : nucleosome-remodeling factor ; ISWI : imitation SWItch ; DREF : DRE-binding factor.
TRF2/TLF
La fonction de TRF2 a été étudiée in vivo chez Caenorhabditis elegans, Xenopus laevis et le poisson zèbre. Dans ces trois organismes, TRF2 est nécessaire à l’expression d’un sous-ensemble de gènes lors du démarage de la transcription zygotique dans l’embryon précoce, la perte de TRF2 étant létale au stade embryonnaire [20-23]. Le TRF2 maternel est donc nécessaire pour la transcription zygotique de certains gènes essentiels chez ces trois espèces.
TRF2 est également essentiel à l’embryogenèse chez la drosophile où il est indispensable à la progression du cycle cellulaire. La purification du TRF2 endogène à partir d’extraits d’embryons a démontré sa présence dans un complexe multiprotéique [24]. Ce complexe contient des facteurs de remodelage de la chromatine (par exemple la sous-unité catalytique ATPase, IWSI) ainsi que DREF (DNA replication-related element binding factor)(Figure 3C). Chez la drosophile, de nombreux gènes impliqués dans la progression du cycle cellulaire possèdent des éléments (DRE) de fixation pour DREF. TRF2 régule ainsi l’expression de ces gènes en se fixant indirectement sur leur promoteurs par l’intermédiaire de DREF.
L’inactivation de TRF2 dans les cellules lymphoïdes DT40 de poulet a révélé d’autres fonctions [25]. Dans ces cellules, TRF2 est cytoplasmique et est importé dans le noyau uniquement lors de la phase G2 du cycle cellulaire ou après exposition au stress (UV). Par conséquent, les cellules trf2-/- ont une phase G2 raccourcie et sont moins sensibles au stress.
Chez les mammifères, TRF2 a une fonction beaucoup plus spécialisée. Chez la souris, TRF2 est très fortement exprimé dans les cellules méiotiques au stade pachytène et dans les spermatides ronds haploïdes [26]. Les souris trf2-/- sont viables. Contrairement donc aux autres organismes, TRF2 n’est pas essentiel à l’embryogenèse murine. Les souris trf2-/- femelles sont fertiles mais les mâles sont stériles en raison d’une absence totale de spermatozoïdes matures. La spermiogenèse est interrompue par l’apoptose tardive des spermatides ronds. Ces spermatides sont caractérisés par une désorganisation de leur hétérochromatine avec une fragmentation du chromocentre [27].
Ces études démontrent que TRF2 a évolué dans sa fonction : d’un facteur jouant un rôle essentiel pour la viabilité cellulaire et l’embryogenèse, il est devenu un élément hautement spécialisé dans la spermatogenèse.
Du nouveau sur TBP
Des expériences in vitro suggéraient un rôle du domaine aminoterminal de TBP dans la fixation de TBP à l’ADN et la transcription des promoteurs possédant un élément TATA [28]. Chez la levure, la délétion de la région aminoterminale est létale seulement en combinaison avec d’autres mutations dans le domaine conservé affectant la fixation à l’ADN [29]. D’autres études ont mis en évidence une interaction entre le domaine aminoterminal et le complexe SNAPc (small nuclear RNA activating protein complex) qui augmente la transcription du gène U6 par pol III [30]. Malgré ces résultats et des études structurales sur la TBP de Saccharomyces cerevisiae [31], la fonction de ce domaine in vivo restait encore énigmatique.
Inactivation du domaine aminoterminal de TBP
Une mutation astucieuse a été introduite dans le domaine aminoterminal du TBP murin [32]. Ce nouvel allèle code pour une protéine TBP tronquée (TBPΔN), constituée principalement de la région carboxyterminale (Figure 2A). Seul un petit nombre de souris homozygotes pour cette mutation (tbpΔN/ΔN) sont présentes dans la progéniture, mais elles apparaissent normales. Cette observation exclut un rôle essentiel pour le domaine aminoterminal de TBP, conclusion confirmée par l’étude de la transcription des trois ARN polymérases dans les fibroblastes embryonnaires tbpΔN/ΔN [33]. La majorité des embryons tbpΔN/ΔN meurent in utero, mais ne présentent pas de malformations évidentes. En revanche, les placentas sont le siège d’anomalies impliquant le mélange du sang foetal et maternel et d’une abondante hémophagocytose par les cellules géantes du trophoblaste. Par ailleurs, les embryons tbpΔN/ΔN survivent lorsqu’ils se développent dans des souris immunodéprimées, par exemple rag1-/-ou des souris SCID. De plus, la combinaison de la mutation tbpΔN/ΔN∈et de la mutation du gène codant pour la β2-microglobuline (β2m) permet également une viabilité accrue des embryons tbpΔN/ΔN/β2m-/-. Ces expériences indiquent que la mortalité des embryons tbpΔN/ΔNest due à une anomalie du placenta impliquant un défaut de l’établissement de la tolérance foeto-maternelle. L’ensemble de ces observations suggère que le domaine aminoterminal de TBP joue un rôle dans un processus dépendant de la β2m qui normalement inhibe le rejet immunologique maternel du foetus. Le mécanisme moléculaire de cet effet reste inconnu.
Inactivation totale du gène tbp
Le gène codant pour TBP a été inactivé dans des cellules de poulet et chez la souris. L’inactivation d’un des allèles de TBP dans les cellules DT40 provoque un ralentissement du cycle cellulaire et une augmentation de l’apoptose [34] dus à une baisse de l’expression de la phosphatase Cdc25B entraînant une augmentation de la forme phosphorylée et inactive de Cdc2.
Un rôle direct pour TBP dans la régulation du cycle cellulaire est également suggéré par l’observation selon laquelle le niveau de TBP est augmenté dans les cellules transformées par l’oncogène Ras [35]. De plus, la surexpression de TBP provoque la transformation oncogénique. Ces observations démontrent que TBP règle préférentiellement des gènes impliqués dans la progression du cycle cellulaire.
Chez la souris, les animaux TBP hétérozygotes sont normaux et fertiles, mais après croisement, aucune souris tbp-/- n’est détectée. Dans les embryons tbp-/-, la prolifération cellulaire s’arrête entre le stade huit cellules et blastocyste avec la déplétion du TBP maternel [36]. Les blastocystes tbp-/- mis en culture meurent par apoptose après 48 heures. Or, malgré l’impossibilité de détecter TBP dans ces blastocystes, une abondante transcription par pol II, comparable aux cellules sauvages, est observée (Figure 4). En revanche, il y a bien un arrêt de la transcription par les ARN pol I et pol III.
Figure 4
Immunodétection de TBP et de la transcription pol II dans des blastocystes.
A. Immunodétection de TBP dans les blastocystes de souris sauvages (WT) et tbp-/-. Marquage immunofluorescent de blastocystes de souris par un anticorps monoclonal anti-TBP. TBP est visible dans le noyau de chaque cellule des blastocystes tbp+/+ (WT), mais pas dans les blastocystes tbp-/-. Le marquage de l’ADN nucléaire par le colorant Hoechst indique la présence d’un nombre réduit de cellules dans les blastocystes tbp-/-. B. Immunodétection de la transcription pol II dans les blastocystes sauvages et tbp-/-. Après 24 heures de culture, les chaînes d’ARN en cours de synthèse sont marquées par l’incorporation de Br-UTP. L’ARN ainsi marqué est détecté par une réaction croisée avec un anticorps anti-Br-UTP. Les images montrent une cellule individuelle visualisée à l’intérieur d’un blastocyste par microscopie confocale. Un fort marquage du nucléoplasme est visible dans les cellules sauvages et tbp-/- correspondant à la transcription pol II (voir [36]).
Comment expliquer la persistance de la transcription pol II dans les cellules tbp-/-? Il est possible que TRF3 puisse se substituer à TBP. Le domaine carboxyterminal de TRF3 possède une forte similarité avec TBP, et TRF3 fixe les éléments TATA [17]. Néanmoins, il reste à démontrer l’expression de TRF3 et son activité transcriptionnelle dans l’embryon précoce.
Une autre possibilité est que TBP soit nécessaire à l’activation de novo de la transcription, mais pas à la ré-initiation. Une activation de novo a lieu lors du démarrage de la transcription zygotique et après chaque mitose. En effet, TBP reste associé à certains promoteurs dans la chromatine condensée pendant la mitose, programmant ainsi ces promoteurs à une activation précoce au cours du cycle cellulaire [37]. Par conséquent, il est possible que TBP initie la transcription dans les cellules tbp-/- jusqu’à ce que sa concentration devienne insuffisante. TBP est alors remplacé dans le complexe de réinitiation par TRF3 ou un autre facteur permettant une persistance de la transcription pol II.
Il est également possible que cette transcription (initiation et/ou ré-initiation) soit assurée par le complexe TFTC (TBP-free TAFII-containing complex). Le complexe TFTC est constitué d’un sous-ensemble de TAF, mais ne contient pas de TBP [38]. Ce complexe peut se substituer à TFIID et permettre une transcription basale et régulée in vitro. Toutefois, ni TRF3, ni TFTC ne pourraient assurer seuls la transcription de tous les gènes nécessaires à la prolifération cellulaire.
Conclusions
Les résultats présentés ici révèlent des propriétés inattendues pour TRF2 et TBP. D’un élément nécessaire lors de l’embryogenèse, TRF2 est devenu un facteur spécialisé dans la spermatogenèse chez les mammifères. TBP n’est plus à considérer comme un facteur universel de la transcription mais comme un facteur intervenant lors de l’activation de gènes spécifiques contrôlant le cycle cellulaire. De plus, TBP semble posséder une fonction spécialisée liée à son domaine aminoterminal dans l’établisssement de la tolérance foetomaternelle.
Appendices
Remerciements
Nous remercions G. Mengus pour une lecture critique du manuscrit. Le travail de notre laboratoire est soutenu par le CNRS, l’Inserm, l’Université Louis Pasteur, l’Hôpital Universitaire de Strasbourg, le Ministère de la Recherche et de la Technologie, l’Association pour la Recherche contre le Cancer, l’Association Française contre les Myopathies et la Ligue Nationale contre le Cancer.
Références
- 1. Albright SR, Tjian R. TAFs revisited : More data reveal new twists and confirm old ideas. Gene 2000 ; 242 : 1-13.
- 2. Gangloff Y, Romier C, Thuault S, et al. The histone fold is a key structural motif of transcription factor TFIID. Trends Biochem Sci 2001 ; 26 : 250-7.
- 3. Timmers HTM, Meyers RE, Sharp PA. Composition of transcription factor B-TFIID. Proc Natl Acad Sci USA 1992 ; 89 : 8140-4.
- 4. van der Knaap JA, Borst JW, van der Vliet PC, et al. Cloning of the cDNA for the TATA-binding protein-associated factorII170 subunit of transcription factor B-TFIID reveals homology to global transcription regulators in yeast and Drosophila. Proc Natl Acad Sci USA 1997 ; 94 : 11827-32.
- 5. Hernandez N. TBP, a universal eukaryotic transcription factor? Genes Dev 1993 ; 7 : 1291-308.
- 6. Burley SK, Roeder RG. Biochemistry and structural biology of transcription factor IID (TFIID). Annu Rev Biochem 1996 ; 65 : 769-99.
- 7. Burley SK. Picking up the TAB. Nature 1996 ; 381 : 112-3.
- 8. Burley SK. X-ray crystallographic studies of eukaryotic transcription initiation factors. Philos Trans R Soc Lond B Biol Sci 1996 ; 351 : 483-9.
- 9. Liu D, Ishima I, Tong KI, et al. Solution structure of a TBP-TAFII230 complex : Protein mimicry of the minor groove surface of the TATA box unwound by TBP. Cell 1998 ; 94 : 573-83.
- 10. Kamada K, Shu F, Chen H, et al. Crystal structure of negative cofactor 2 recognizing the TBP-DNA transcription complex. Cell 2001 ; 106 : 71-81.
- 11. Dantonel JC, Wurtz JM, Poch O, et al. The TBP-like factor : An alternative transcription factor in metazoa? Trends Biochem Sci 1999 ; 24 : 335-9.
- 12. Crowley TE, Hoey T, Liu JK, et al. A new factor related to TATA-binding protein has highly restricted expression patterns in Drosophila. Nature 1993 ; 361 : 557-61.
- 13. Rabenstein MD, Zhou S, Lis JT, Tjian R. TATA box-binding protein (TBP)-related factor 2 (TRF2), a third member of the TBP family. Proc Natl Acad Sci USA 1999 ; 96 : 4791-6.
- 14. Teichmann M, Wang Z, Martinez E, et al. Human TATA-binding protein-related factor-2 (hTRF2) stably associates with hTFIIA in HeLa cells. Proc Natl Acad Sci USA 1999 ; 96 : 13720-5.
- 15. Moore PA, Ozer J, Salunek M, et al. A human TATA binding protein-related protein with altered DNA binding specificity inhibits transcription from multiple promoters and activators. Mol Cell Biol 1999 ; 19 : 7610-20.
- 16. Hansen SK, Takada S, Jacobson RH, et al. Transcription properties of a cell type-specific TATA-binding protein, TRF. Cell 1997 ; 91 : 71-83.
- 17. Persengiev SP, Zhu X, Dixit BL, et al. TRF3, a TATA-box-binding protein-related factor, is vertebrate-specific and widely expressed. Proc Natl Acad Sci USA 2003 ; 100 : 14887-91.
- 18. Holmes MC, Tjian R. Promoter-selective properties of the TBP-related factor TRF1. Science 2000 ; 288 : 867-70.
- 19. Takada S, Lis JT, Zhou S, Tjian R. A TRF1/BRF complex directs Drosophila RNA polymerase III transcription. Cell 2000 ; 101 : 459-69.
- 20. Dantonel JC, Quintin S, Lakatos L, et al. TBP-like factor is required for embryonic RNA polymerase II transcription in C. elegans. Mol Cell 2000 ; 6 : 715-22.
- 21. Kaltenbach L, Horner MA, Rothman JH, Mango SE. The TBP-like factor CeTLF is required to activate RNA polymerase II transcription during C. elegans embryogenesis. Mol Cell 2000 ; 6 ; 705-13.
- 22. Veenstra GJ, Weeks DL, Wolffe AP. Distinct roles for TBP and TBP-like factor in early embryonic gene transcription in xenopus. Science 2000 ; 290 : 2312-5.
- 23. Muller F, Lakatos L, Dantonel J, et al. TBP is not universally required for zygotic RNA polymerase II transcription in zebrafish. Curr Biol 2001 ; 11 : 282-7.
- 24. Hocheimer A, Zhou S, Zheng S, et al. TRF2 associates with DREF and directs promoter-selective gene expression in Drosophila. Nature 2002 ; 420 : 439-45.
- 25. Shimada M, Nakadai T, Tamura TA. TATA-binding protein-like protein (TLP/TRF2/TLF) negatively regulates cell cycle progression and is required for the stress-mediated G(2) checkpoint. Mol Cell Biol 2003 ; 23 : 4107-20.
- 26. Martianov I, Fimia GM, Dierich A, et al. Late arrest of spermiogenesis and germ cell apoptosis in mice lacking the TBP-like TLF/TRF2 gene. Mol Cell 2001 ; 7 : 509-15.
- 27. Martianov I. Brancorsini S, Gansmuller A, et al. Distinct functions of TBP and TLF/TRF2 during spermatogenesis : Requirement of TLF for heterochromatic chromocenter formation in haploid round spermatids. Development 2002 ; 129 : 945-55.
- 28. Lescure A, Lutz Y, Eberhardt D, et al. The N-terminal domain of the human TATA-binding protein plays a role in transcription from TATA-containing RNA polymerase II and III promoters. EMBO J 1994 ; 13 : 1166-75.
- 29. Lee M, Struhl K. Multiple functions of the nonconserved N-terminal domain of yeast TATA- binding protein. Genetics 2001 ; 158 : 87-93.
- 30. Mittal V, Hernandez N. Role for the amino-terminal region of human TBP in U6 snRNA transcription. Science 1997 ; 275 : 1136-40.
- 31. Khrapunov S, Pastor N, Brenowitz M. Solution structural studies of the Saccharomyces cerevisiae TATA binding protein (TBP). Biochemistry 2002 ; 41 : 9559-71.
- 32. Hobbs NK, Bondareva AA, Barnett S, et al. Removing the vertebrate-specific TBP N terminus disrupts placental β2m-dependent interactions with the maternal immune system. Cell 2002 ; 110 : 43-54.
- 33. Schmidt EE, Bondareva AA, Radke JR, Capecchi MR. Fundamental cellular processes do not require vertebrate-specific sequences within the TATA-binding protein. J Biol Chem 2003 ; 278 : 6168-74.
- 34. Um M, Yamauchi J, Kato S, Manley JL. Heterozygous disruption of the TATA-binding protein gene in DT40 cells causes reduced cdc25B phosphatase expression and delayed mitosis. Mol Cell Biol 2001 ; 21 : 2435-48.
- 35. Johnson SA, Dubeau L, Kawalek M, et al. Increased expression of TATA-binding protein, the central transcription factor, can contribute to oncogenesis. Mol Cell Biol 2003 ; 23 : 3043-51.
- 36. Martianov I, Viville S, Davidson I. RNA polymerase II transcription in murine cells lacking the TATA binding protein. Science 2002 ; 298 : 1036-9.
- 37. Christova R, Oelgeschlager T. Association of human TFIID-promoter complexes with silenced mitotic chromatin in vivo. Nat Cell Biol 2002 ; 4 : 79-82.
- 38. Wieczorek E, Brand M, Jacq X, Tora L. Function of TAFII-containing complex without TBP in transcription by RNA polymerase II. Nature 1998 ; 393 : 187-91.
List of figures
Figure 1
Structure des promoteurs des gènes transcrits par les ARN polymérases I, II et III.
TBP (TATA binding protein) est une sous-unité des complexes nécessaires à la transcription par les trois ARN polymérases. A. La transcription par l’ARN polymérase I (Pol I) des gènes codant pour les ARN ribosomiques (ARNr) nécessite la fixation de UBF (upstream binding factor) à l’élément régulateur UE (upstream element) et la fixation du complexe SL1 (selectivity factor 1), contenant TBP et trois TAF (TBP-associated factors), au promoteur proximal CE (core element). B. La transcription par l’ARN polymérase II (pol II) des ARNm peut faire intervenir le complexe TFIID (transcription factor IID), formé de TBP et de treize TAF (représentés ici par un seul disque). C. La transcription par pol II peut aussi se faire par l’intermédiaire du complexe B-TFIID formé de TBP et BTAF1. D. TRF3 (TBP-related factor) est un nouveau facteur impliqué dans la transcription par pol II. Son interaction avec les TAF reste à démontrer. E. La transcription par pol III fait intervenir les complexes TFIIIA et TFIIIC qui se fixent sur les éléments intragéniques, par exemple du gène codant pour les ARN ribosomiques 5S (ARN 5S) comme indiqué ici. Le complexe TFIIIB contenant TBP se fixe en amont du gène. F. Dans le cas de la transcription du gène codant pour les petits ARN nucléaires U6 (ARN U6), TBP se fixe sur l’élément TATA et interagit avec le complexe SNAPc (small nuclear RNA-activating protein complex) fixé sur PSE (proximal sequence element) par l’intermédiaire de son domaine aminoterminal. L’activation du gène ARN U6 se fait par le facteur Oct1 (octamer-binding transcription factor 1) fixé à une séquence OCTA (octamer). Pour plus de clarté, les cinq sous-unités du complexe SNAPc sont réprésentées par un seul disque. BRF2 : TFIIB-related factor, subunit of RNA polymerase III transcription initiation factor.
Figure 2
Organisation et structure de la famille des TBP (TATA binding proteins).
A. Organisation de TBP de différentes espèces. Le domaine aminoterminal est réprésenté en jaune et la présence des répétitions des résidus glutamines (Q) en noir. Le polymorphisme allélique humain (28-42 glutamines) est indiqué. La structure du mutant TBPΔN murin (souris ³N) mentionné dans l’article ainsi que l’épitope FLAG (étiquette génétique) (F) sont également schématisés. L’alignement de TRF1 (TBP-related factor 1) et de la famille TBP sont présentés. Le premier acide aminé du domaine conservé est indiqué pour chacun des TBP. B, C. Alignement de TRF3 et TRF2/TLF (TBP-like factor) et de la famille TBP. La répétition directe qui forme la structure en selle de cheval du domaine conservé est indiquée par les flèches. D. Représentation de la structure en selle de cheval du domaine carboxyterminal conservé de TBP, déterminée par cristallographie aux rayons X. H1, H1’, H2 et H2’ représentent les hélices α et S1-S5 et S1’-S5’ les feuillets β.
Figure 3
Transcription par TRF1 et TRF2 (TBP-related factors 1 et 2).
A. La structure du promoteur du gène Tudor est schématisée ainsi que les complexes qui interviennent à chaque site d’initiation. B. Schéma du gène codant pour les ARN ribosomiques 5S (ARN 5S) (voir Figure 1E) sous le contrôle du complexe TRF1-BRF (TFIIB-related factor). C. La structure du promoteur du gène PCNA(proliferating cell nuclear antigen) est schématisée montrant la présence des sites de fixation TATA, E2F (facteur de transcription E2F) et DRE (DNA replication-related element). Le complexe TFIID (transcription factor IID) initie la transcription au promoteur proximal et le complexe TRF2 au promoteur distal, une organisation analogue à la transcription de Tudor à partir de deux sites d’initiation par TFIID et TRF1. TAF : TBP-associated factor ; NURF : nucleosome-remodeling factor ; ISWI : imitation SWItch ; DREF : DRE-binding factor.
Figure 4
Immunodétection de TBP et de la transcription pol II dans des blastocystes.
A. Immunodétection de TBP dans les blastocystes de souris sauvages (WT) et tbp-/-. Marquage immunofluorescent de blastocystes de souris par un anticorps monoclonal anti-TBP. TBP est visible dans le noyau de chaque cellule des blastocystes tbp+/+ (WT), mais pas dans les blastocystes tbp-/-. Le marquage de l’ADN nucléaire par le colorant Hoechst indique la présence d’un nombre réduit de cellules dans les blastocystes tbp-/-. B. Immunodétection de la transcription pol II dans les blastocystes sauvages et tbp-/-. Après 24 heures de culture, les chaînes d’ARN en cours de synthèse sont marquées par l’incorporation de Br-UTP. L’ARN ainsi marqué est détecté par une réaction croisée avec un anticorps anti-Br-UTP. Les images montrent une cellule individuelle visualisée à l’intérieur d’un blastocyste par microscopie confocale. Un fort marquage du nucléoplasme est visible dans les cellules sauvages et tbp-/- correspondant à la transcription pol II (voir [36]).