Article body
Un certain nombre de tissus, dont la peau, l’intestin et le système hématopoïétique se renouvellent en permanence, même à l’âge adulte. D’autres tissus, comme le foie ou le muscle, ne se renouvellent que lorsqu’ils ont été lésés. L’homéostasie et la régénération tissulaires dans ces situations reposent sur l’existence de cellules particulières appelées «cellules souches» ((→) m/s 2003, n° 6-7, p.683). Les cellules souches partagent un ensemble de propriétés commu-nes. Elles se renouvellent, c’est-à-dire qu’au moins une des cellules filles possède les mêmes caractéristiques que la cellule souche initiale, et elles ont la possibilité de se différencier en cellules matures qui composent leur tissu d’origine.
Les cellules souches résident dans un micro-environnement particulier appelé «niche» [1] ((→) m/s 2004, n° 2, p.156). Cette niche comprend les cellules souches elles-mêmes, des cellules de soutien, des facteurs solubles et la proximité cellulaire favorise les interactions des cellules entre elles et avec les matrices extracellulaires. L’ensemble de ces différents facteurs permet de contrôler la prolifération, la migration et la différenciation des cellules souches.
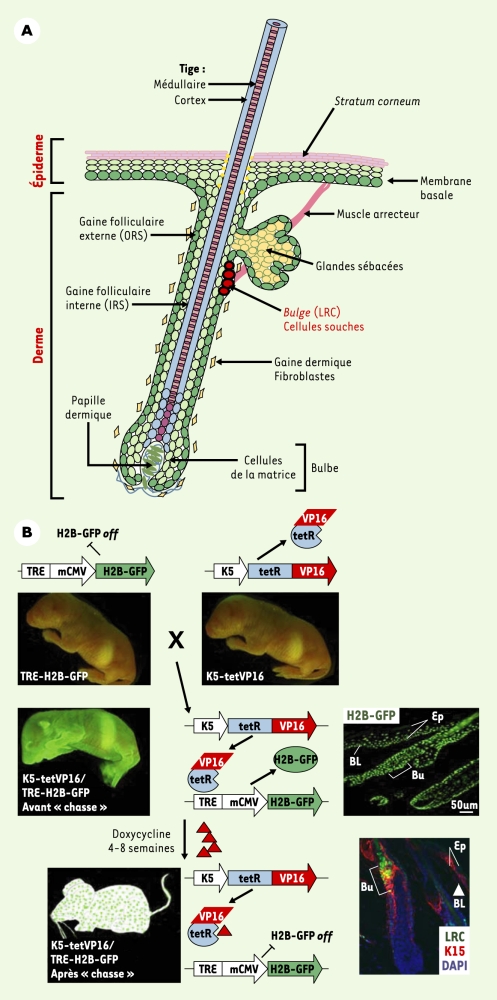
Les cellules souches épithéliales se divisent plus rarement que leurs descendantes. Cette caractéristique a été utilisée pour localiser les cellules souches dans le tissu d’origine. L’administration continue (pulse) d’analogues de nucléotides (BrdU, bromodésoxyuridine, 3H-thymidine, thymidine tritiée) qui s’intègrent dans l’ADN en phase de réplication, permet de marquer l’ensemble des cellules ayant traversé la phase S du cycle cellulaire, y compris les cellules souches. Après une période d’attente (chase) plus ou moins longue après le sevrage en BrdU, la plupart des cellules en phase active de prolifération perdent leur marquage par dilution (chassent le BrdU) et seulement les cellules souches qui se sont divisées plus lentement, retiennent le marquage et sont appelées pour cette raison en anglais label-retaining cells ou LRC [2]. L’application de cette technique à la caractérisation de la hiérarchie cellulaire dans le tissu cutané et ses annexes a permis de montrer que les «LRC» de la peau sont confinées au niveau du bulge, une portion spécialisée d’une des enveloppes épidermiques du follicule pileux [3] (Figure 1A). Les cellules souches du bulge sont multipotentes [4, 5]. Les précurseurs qui en sont issus donneront naissance à l’ensemble des types cellulaires (plus d’une dizaine) qui constitueront le poil. Dans certaines circonstances, comme une plaie cutanée, les cellules souches du bulge peuvent aussi migrer vers l’épiderme blessé et contribuer à la cicatrisation [6]. Jusqu’à présent, il n’était pas possible d’isoler les «LRC» vivantes de leur tissu afin d’étudier ex vivo leurs caractéristiques. Pour cette raison, notre équipe a développé une nouvelle technique de pulse and chase qui permet de marquer et d’isoler spécifiquement les cellules souches du bulge [7]. Cette stratégie nécessite l’expression de deux transgènes différents dans une même souris: un premier transgène code pour une protéine nucléaire, l’histone H2B fusionnée à la green fluorescent protein (H2B-GFP), sous le contrôle d’un promoteur inductible par la tétracycline (TRE-H2B-GFP) (Figure 1B). Le second transgène code pour le facteur de transcription Tétracycline Tet-VP16 sous le contrôle du promoteur de la kératine 5 (K5) [8], et le croisement des deux souris permet l’expression de l’histone H2B-GFP dans l’ensemble des cellules épidermiques, y compris dans les cellules du bulge(Figure 1B) ((→) m/s 2003, n°5, p.755). C’est l’équivalent d’une période d’incorporation d’un analogue nucléotidique ou pulse. Si l’on ajoute la doxycycline, un analogue de la tétracycline, l’expression de H2B-GFP est réprimée. Les cellules en division active vont perdre la GFP par dilution (équivalent de la période chase); mais le produit du transgène H2B-GFP est très stable, et seules les cellules se divisant rarement, présentes dans le bulge, restent marquées par la GFP (Figure 1B). Cette stratégie nous a permis d’isoler par cytométrie de flux ces cellules exprimant la GFP, vivantes; nous avons ensuite défini le profil transcriptionnel de ces LRC du bulge et nous l’avons comparé à celui des cellules en voie de différenciation localisées à l’extérieur de la niche épithéliale. La liste des gènes différemment exprimés est disponible sur la toile à l’adresse suivante:www.rockefeller.edu/labheads/ fuchs/database.php.
Figure 1
A. Follicule pileux en anagène. Un follicule pileux est une structure complexe formée d’une gaine mésenchymateuse en-tourant plusieurs gaines de cellules épithéliales (gaine épithéliale externe/ORS et interne/IRS). La membrane basale de l’épiderme est en continuité avec la gaine épithéliale externe de l’épiderme. Les cellules souches sont localisées dans un renflement ou bulge situé sous la glande sébacée. La membrane basale, les cellules de la gaine folliculaire externe (ORS) et le bulge expriment la kératine 5. B. Stratégie pour marquer les cellules souches épitheliales peu proliférantes in vivo [7]. Cette stratégie est fondée sur un système inductible, répressible par la tétracycline (Tet-off). Des souris double transgéniques ont été produites, exprimant: (1) une histone H2B-GFP sous le contrôle d’un élément de réponse à la tétracycline (TRE); (2) un activateur transcriptionnel réglé par la tétracycline sous le contrôle du promoteur de la kératine 5 (K5-TetR-VP16). En l’absence de doxycycline (un analogue de la tétracycline), les souris ayant les deux transgènes expriment le marqueur GFP dans la membrane basale, l’ORS et le bulge. Un exemple de coupe de peau est représenté montrant en vert l’expression de l’histone H2B-GFP. En présence de doxycycline, l’activateur transcriptionnel K5-TetR-VP16 est réprimé, le marqueur GFP ne s’exprime plus et les cellules en division perdent tout marquage au bout de 4 à 8 semaines. Seules les cellules quiescentes expriment la GFP au niveau de leur noyau. Une coupe de peau est représentée montrant en vert les LRC du bulge et en rouge un immunomarquage pour la kératine 15 localisé dans le bulge et la membrane basale. Un marqueur d’ADN (DAPI) colore le noyau des cellules en bleu. BL: membrane basale. Ep: épiderme; Bu: bulge.
Certains ARNm, surexprimés dans les LRC, sont également présents dans des cellules souches provenant d’autres tissus (cellules souches hématopoïétiques de la moelle osseuse, cellules issues de neurosphères, cellules souches embryonnaires ES) [9, 10] alors que d’autres ARNm définissent un environnement propre à la niche des cellules souches du follicule pileux. Il est intéressant de noter que 43% des ARNm spécifiquement surexprimés dans les cellules souches du bulge codent pour des protéines sécrétées ou membranaires. Cette analyse transcriptionnelle révèle que les cellules souches présentes dans leur niche sont différentes de leurs descendantes. Le devenir des cellules souches va dépendre de la présence de facteurs intrinsèques à ces cellules (facteurs de transcription, protéines contrôlant le cycle cellulaire...) mais aussi de leur capacité à répondre à des facteurs extrinsèques présents au sein de la niche. Notre étude montre que les cellules souches du bulge participent activement à l’élaboration de cette niche. Elles expriment à leur surface certains récepteurs, FZD2 par exemple, qui leur permettent de répondre à des facteurs solubles et d’autres, comme l’intégrine∈β6, qui leur permettent de se lier à la matrice extracellulaire (Figure2). Elles sécrètent également des protéines solubles qui activent ou modulent des voies de signalisation comme la voie Wnt ou TGF-β. Ces cellules sécrètent aussi des composants de leur matrice extracellulaire comme la ténascine C ((→) m/s 2003, n°4, p.443) ou certains sous-types de collagène. Les cellules du bulge pourraient donc avoir la capacité d’organiser leur niche, de communiquer avec les cellules voisines et ainsi de pouvoir répondre à leur environnement (Figure2). Nous poursuivons maintenant l’étude du rôle de ces différents gènes dans la biologie des cellules souches épithéliales.
Figure 2
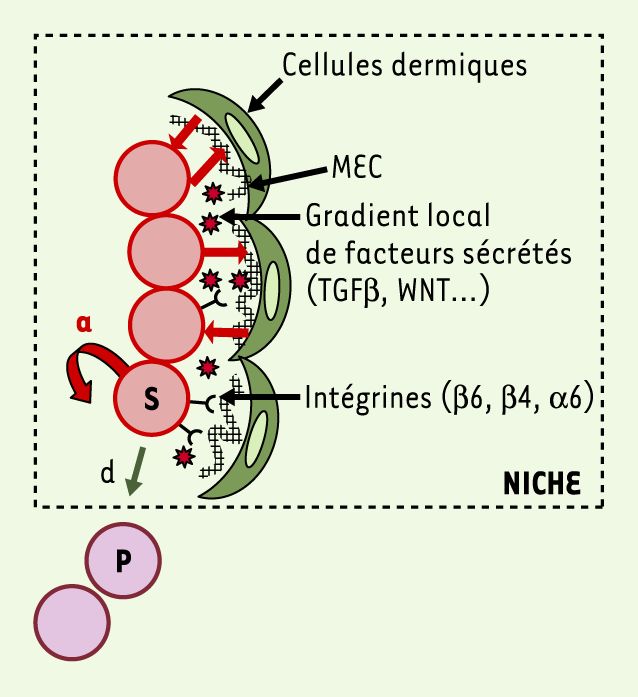
Un modèle de cellules souches dans leur niche.
Les cellules souches (S) dans le bulge s’autorenouvellent (a) pour maintenir un pool constant de cellules souches et se différencient (d) en cellules progénitrices (P) qui quittent le bulge. Les cellules du bulge interagissent avec les composants de la matrice extracellulaire (MEC) (grillagé noir) qui permettent probablement de séquestrer et de moduler la concentration locale des facteurs sécrétés par les cellules souches.
Cette nouvelle technique qui réalise l’équivalent d’un pulse and chase adapté à un gène codant pour une protéine fluorescente, devrait aussi permettre d’étudier et d’isoler les cellules souches de bien d’autres tissus (comme les cellules souches neurales, ou intestinales) pour lesquelles il n’existe pas encore de marqueur spécifique.
Appendices
Remerciements
Ces travaux ont été menés dans le Laboratoire de Elaine Fuchs à l’Université de Rockefeller, New York. GG et CB sont financés par Human Frontier Science Program et par la Fondation Belgian American Foundation et OTAN (CB).
Références
- 1. Schofield R. The relationship between the spleen colony-forming cell and the haematopoietic stem cell. Blood Cells 1978; 4: 7-25.
- 2. Bickenbach JR. Identification and behaviour of label-retaining cells in oral mucosa and skin. J Dent Res 1981; 60 (n°spécial): 1611-20.
- 3. Cotsarelis G, Sun TT, Lavker RM. Label-retaining cells reside in the bulge area of pilosebaceous unit: implications for follicular stem cells, hair cycle, and skin carcinogenesis. Cell 1990; 61: 1329-37.
- 4. Oshima H, Rochat A, Kedzia C, et al. Morphogenesis and renewal of hair follicles from adult multipotent stem cells. Cell 2001; 104: 233-45.
- 5. Taylor G, Lehrer MS, Jensen PJ, et al. Involvement of follicular stem cells in forming not only the follicle but also the epidermis. Cell 2000; 102: 51-61.
- 6. Alonso L, Fuchs E. Stem cells of the skin epithelium. Proc Natl Acad Sci USA 2003; 100(suppl1): 11830-5.
- 7. Tumbar D, Guasch G, Greco V, et al. Defining the epithelial stem cell niche in skin. Science 2004; 303: 359-63.
- 8. Diamond I, Owolabi T, Marco M. Conditional gene expression in the epidermis of transgenic mice using the tetracycline-regulated transactivators tTA and rTA linked to the keratin 5 promoter. J Invest Dermatol 2000; 115: 788-94.
- 9. Ivanova NB, Dimos JT, Schaniel C, et al. A stem cell molecular signature. Science 2002; 298: 601-4.
- 10. Ramalho-Santos M, Yoon S, Matsuzaki Y, et al. «Stemness»: transcriptional profiling of embryonic and adult stem cells. Science 2002; 298: 597-600.
List of figures
Figure 1
A. Follicule pileux en anagène. Un follicule pileux est une structure complexe formée d’une gaine mésenchymateuse en-tourant plusieurs gaines de cellules épithéliales (gaine épithéliale externe/ORS et interne/IRS). La membrane basale de l’épiderme est en continuité avec la gaine épithéliale externe de l’épiderme. Les cellules souches sont localisées dans un renflement ou bulge situé sous la glande sébacée. La membrane basale, les cellules de la gaine folliculaire externe (ORS) et le bulge expriment la kératine 5. B. Stratégie pour marquer les cellules souches épitheliales peu proliférantes in vivo [7]. Cette stratégie est fondée sur un système inductible, répressible par la tétracycline (Tet-off). Des souris double transgéniques ont été produites, exprimant: (1) une histone H2B-GFP sous le contrôle d’un élément de réponse à la tétracycline (TRE); (2) un activateur transcriptionnel réglé par la tétracycline sous le contrôle du promoteur de la kératine 5 (K5-TetR-VP16). En l’absence de doxycycline (un analogue de la tétracycline), les souris ayant les deux transgènes expriment le marqueur GFP dans la membrane basale, l’ORS et le bulge. Un exemple de coupe de peau est représenté montrant en vert l’expression de l’histone H2B-GFP. En présence de doxycycline, l’activateur transcriptionnel K5-TetR-VP16 est réprimé, le marqueur GFP ne s’exprime plus et les cellules en division perdent tout marquage au bout de 4 à 8 semaines. Seules les cellules quiescentes expriment la GFP au niveau de leur noyau. Une coupe de peau est représentée montrant en vert les LRC du bulge et en rouge un immunomarquage pour la kératine 15 localisé dans le bulge et la membrane basale. Un marqueur d’ADN (DAPI) colore le noyau des cellules en bleu. BL: membrane basale. Ep: épiderme; Bu: bulge.
Figure 2
Un modèle de cellules souches dans leur niche.
Les cellules souches (S) dans le bulge s’autorenouvellent (a) pour maintenir un pool constant de cellules souches et se différencient (d) en cellules progénitrices (P) qui quittent le bulge. Les cellules du bulge interagissent avec les composants de la matrice extracellulaire (MEC) (grillagé noir) qui permettent probablement de séquestrer et de moduler la concentration locale des facteurs sécrétés par les cellules souches.