Article body
Depuis des décennies, les relations de parenté entre l’homme et les grands singes (chimpanzés, gorilles et orangs-outans) suscitent de nombreux débats, controverses et une pléthore de publications ; deux faits principaux ont longtemps agité la communauté scientifique des primatologues. Dans les années 1960, alors que la classification tra-ditionnelle restreignait la famille Hominidae à l’homme (les grands singes africains et asiatiques étant regroupés dans la famille des Pongidae), l’analyse de certaines protéines sérologiques suggérait l’existence d’un ancêtre commun à l’homme et au chimpanzé, qui excluait le gorille [1]. Le chimpanzé était donc plus proche de l’homme qu’il ne l’était du gorille, ce que confirma la grande ressemblance génétique (98 % d’homologie) entre l’homme (Homo sapiens) et le chimpanzé commun (Pan troglodytes) [2]. Ces différents travaux, et bien d’autres, conduisirent à regrouper dans la famille Hominidae l’homme et les grands singes africains (chimpanzés et gorilles), les orangs-outans devenant les seuls représentants actuels de la famille Pongidae.
Si la ressemblance génétique entre Homo et Pan, estimée à 98-99 %, a été au centre du débat pendant longtemps, la question de la différence génétique, donc des 1 à 2 %, s’y est insinuée plus récemment. En effet, ce sont bien ces 1,4 % de différence qui font que l’homme est homme, et les recherches se sont orientées à la fin du xxe siècle dans cette direction [3]. Ces premiers résultats montraient par exemple que les cellules humaines avaient perdu une forme particulière de l’acide sialique (acide N-acétyl-neuraminique), présent à la surface de toutes les cellules, et jouant un rôle dans la transmission de pathogènes comme le choléra, la grippe, la malaria… maladies auxquelles les chimpanzés sont moins sensibles que l’homme, ce qui pourrait être le fait de la modification de cette molécule. À partir des années 2000, la comparaison des génomes humain et de grands singes a confirmé la faible di-vergence génétique - évaluée à 1,2 %-1,75 % selon le type de données [4-9], voire 0,6 % pour des sites non synonymes [10] - entre l’homme et le chimpanzé commun. Le temps de divergence (ancêtre commun) est alors estimé à 4,6-6,2 millions d’années (MA) pour l’homme et les chimpanzés, et à 6,2-8,4 MA pour le gorille [5].
Par ailleurs, le séquençage du génome complet de chimpanzé commun[1], débuté dès 1998, vient de se terminer, et une version préliminaire est désormais publiée et accessible sur Internet (GenBank). Une comparaison des deux génomes - chimpanzé et homme - est également effectuée par une équipe internationale de l’université Washington de Seattle (USA). L’alignement des deux séquences devrait faciliter la recherche biomédicale en permettant d’identifier grossièrement les différences et les similitudes, peut-être indicatives des changements biologiques survenus au cours de l’évolution, depuis la divergence entre l’homme et les chimpanzés.
Très récemment, en décembre 2003, une équipe américaine (un consortium public-privé) dirigée par Andrew Clark de l’université Cornell à New York, et Michelle Cargill de la firme de biotechnologie Celera Diagnostics en Californie a publié dans la revue Science la comparaison pour l’homme, le chimpanzé commun et la souris (représentative de l’espèce non primate nécessaire à la comparaison) de 7 645 gènes [11]. La classification en grandes fonctions biologiques et en familles de gènes est établie avec le système PANTHER. Sur les 200 000 sé-quences initiales, 7 645, pour lesquelles l’orthologie a pu être confirmée, ont été retenues. Elles ont ensuite été soumises à différents modèles et tests statistiques incluant de nombreux paramètres afin d’identifier les gènes pour lesquels une évolution accélérée due à une pression de sélection positive (avantage sélectif), et non pas au hasard (dérive génique), est mise en évidence, et ce pour chacune des trois lignées. Cette pression de sélection positive caractérise 1 547 gènes humains et 1 534 gènes de chimpanzés et, pour nombre d’entre eux, les évolutions - et donc les pressions de sélection - divergeaient entre les trois espèces. Si de nombreux gènes montrent des valeurs non significatives, ceux contrôlant certaines fonctions biologiques présentent une évolution rapide : audition, catabolisme des acides aminés, processus du développement (squelette) ou de neurogenèse. Chez l’homme, il s’agit majoritairement de gènes impliqués dans l’olfaction (une cinquantaine, presque tous codant pour des récepteurs olfactifs [OR]) et il semblerait donc que le mode de vie différent de l’homme et de son plus proche parent ait conduit à des pressions de sélection différentes pour ces récepteurs. L’olfaction n’étant pas un sens prédominant pour la survie de l’homme, il aurait été délaissé au profit d’autres sens au cours de l’évolution humaine. De même, le gène FOXP2 (forkhead-box P2 transcription factor), impliqué dans le développement du langage (une des caractéristiques définissant l’espèce humaine ?), présente un profil de substitutions inhabituel, comme plusieurs autres gènes (une vingtaine) impliqués dans le développement de l’audition et qui paraissent également subir une évolution adaptative dans la lignée humaine. Une mutation dans le gène FOXP2 conduit à une condition connue nommée SLI (specific language impairment) pouvant notamment engendrer des problèmes d’élocution, des difficultés à former des mots et à comprendre et utiliser la grammaire, donc à une perte des compétences nécessaires au développement du langage. Par ailleurs, le gène ayant la pression de sélection positive la plus significative chez l’homme code pour l’α-tectorine, une protéine qui joue un rôle vital dans la membrane tectoriale de l’oreille interne et dont les anomalies sont responsables de surdité d’origine génétique. Pour les auteurs, la compréhension du langage parlé et son apprentissage ont requis des mises au point de l’acuité auditive.
Les auteurs disposent maintenant d’une liste de gènes qui font que « l’homme est homme », et souhaitent l’utiliser pour mieux comprendre les liens entre fonctions moléculaires et processus biologiques, et comparer la variabilité homme-chimpanzé à celle qui existe au sein de l’espèce humaine. Les perspectives sont donc nombreuses : analyse de gènes de régulation, comparaison entre chromosomes, impact des taux de recombinaison, contenu en GC, différences d’expression des gènes et ajout de génomes de mammifères supplémentaires.
Confirmant la multiplication des travaux dans ce domaine, deux nouvelles études publiées en janvier 2004 complètent la liste des « gènes humains » ; l’une analyse un gène lié à la taille du « cerveau » [12] et l’autre à la vision des couleurs [13].
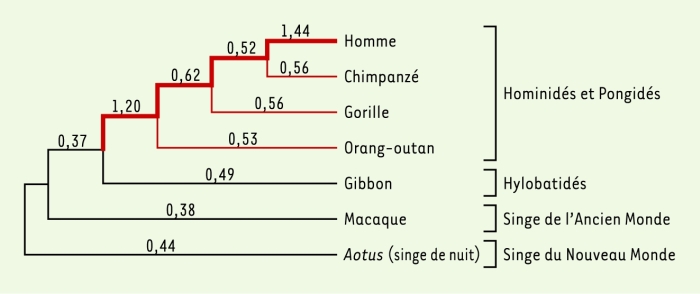
Une autre caractéristique de l’évolution humaine reste en effet l’augmentation progressive de la taille du cortex cérébral. Le gène ASPM (abnormal spindle-like microcephaly associate) joue un rôle important dans ce processus évolutif, sa perte de fonction conduisant à une microcéphalie, c’est-à-dire à une réduction sévère de la taille du cortex cérébral. Bruce Lahn et al., de l’Howard Hughes Medical Institute, ont comparé la séquence de ce gène pour sept espèces de primates, dont l’homme [12] (Figure 1). Pour chaque espèce, les chercheurs ont identifié des mutations responsables (mutations non synonymes) ou non (mutations synonymes) de changements de structure de la protéine codée par ASPM. Seules les premières sont susceptibles d’avoir subi une pression de sélection, les autres reflétant le taux de mutation global. Le rapport entre les deux types de mutations donne une mesure de l’évolution du gène sous la pression de la sélection naturelle. Dans le cas de ASPM, cette analyse conclut à une accélération significative de l’évolution de ce gène dans la lignée menant aux grands singes et à l’homme, par rapport à celle qui est observée pour d’autres primates de l’Ancien Monde (Afrique et Asie) et du Nouveau Monde (les platyrhiniens d’Amérique du Sud) et pour d’autres mammifères (carnivores, rongeurs, artiodactyles). Par ailleurs, l’excès de substitutions non synonymes par rapport aux substitutions synonymes traduit une sélection positive, au moment de la divergence Homo-Pan. L’étude du polymorphisme d’ASPM dans l’espèce humaine suggère qu’un changement avantageux d’acides aminés s’est probablement produit tous les 300 à 400 000 ans. Le gène ASPM a donc subi une évolution adaptative forte chez les grands singes et surtout chez l’homme et son rôle possible dans l’élargissement du cortex cérébral - par exemple par la régulation du nombre de neurones produits par division cellulaire - est envisagé.
Figure 1
Phylogénie du gène ASPM chez les primates.
Les lignées hominidés/pongidés sont surlignées en rouge et celle menant à la lignée humaine en gras. Le rapport taux de substitutions des mutations non-synonymes/mutation synonymes est indiqué sur l’arbre phylogénétique pour chacune des branches (d’après [12]).
En janvier 2004 également, une équipe germano-israélienne dirigée par Y. Gilad [13] a étudié le processus de dégénérescence du répertoire olfactif chez les primates (19 espèces) en estimant la proportion de gènes non fonctionnels pour 100 gènes OR pris au hasard. En effet, la proportion de pseudogènes dans cette famille est d’environ 60 % chez l’homme, 35 % chez les grands singes et moins de 20 % chez les singes du Nouveau Monde et chez d’autres mammifères. L’augmentation du nombre de pseudogènes semble corrélée à la décroissance du sens de l’olfaction pour des espèces chez lesquelles les signaux visuels et auditifs sont devenus plus importants. Une proportion élevée significative de pseudogènes (par rapport à 6 autres platyrhiniens et un lémur) caractérise les grands singes, les cercopithécoïdes et un platyrhinien, le singe hurleur Alouatta : la diminution de taille du répertoire olfactif se serait donc produite deux fois indépendamment au cours de l’évolution des primates. Mais, par ailleurs, l’homme, les grands singes et les singes de l’Ancien Monde possèdent la vision en trois couleurs, tandis que les singes d’Amérique du Sud ne distinguent en général que deux couleurs. L’Alouatta étant le seul singe du Nouveau Monde qui possède une pleine vision trichromatique, les auteurs supposent que la vision trichromatique des primates se serait développée parallèlement à la détérioration du sens de l’olfaction, la vision tricolore compensant la perte d’odorat pour la recherche de nourriture.
Cependant, deux points ne sont jamais abordés dans ce type d’analyse et méritent d’être soulignés. L’homme, Homo sapiens, est le seul représentant actuel du genre Homo ; c’est aussi le cas pour les genres Gorilla et Pongo qui ne comportent qu’une seule espèce et plusieurs sous-espèces ; en revanche, ce n’est pas le cas des chimpanzés. Pour le genre Pan, on distingue deux espèces : Pan troglodytes (le chimpanzé commun avec quatre sous-espèces) et Pan paniscus (le bonobo). Le bonobo, décrit pour la première fois dans la littérature en 1929, a un habitat naturel restreint à une petite zone forestière de la République démocratique du Congo. Il présente des caractéristiques morphologiques et comportementales très particulières qui tendent à le rapprocher de l’homme - notamment son comportement sexuel qui fait régulièrement la « une » des journaux. La quasi-totalité des analyses moléculaires montre que les deux espèces de Pan sont étroitement apparentées avec une divergence estimée à 2-3 MA.
Il est regrettable que l’espèce de chimpanzé concernée par les analyses de comparaison hommes-grands singes publiées ne soit que très rarement précisée. Quant à la sous-espèce de chimpanzés communs, pour lesquels il existe un polymorphisme génétique réel, elle ne l’est évidemment jamais… De plus, les auteurs se concentrent sur les gènes qui font que « l’homme est homme », mais quels sont ceux qui font que le « chimpanzé est chimpanzé » ? En effet, l’ancêtre des chimpanzés et de l’homme n’est pas un chimpanzé ! Il serait donc intéressant dans ce contexte d’étudier les gènes montrant une pression de sélection positive pour identifier les caractéristiques propres aux chimpanzés depuis leur séparation d’avec la lignée humaine, et non pas seulement la condition ancestrale.
Appendices
Note
-
[1]
Ce Primate Genome Project impliquait plusieurs centres de recherche, sous la direction de l’Institut national de recherche sur le génome humain (NHGRI), qui dépend des National Institutes of Health (NIH) basés à Bethesda (Maryland, USA).
Références
- 1. Goodman M. Serological analysis of the systematics of recent hominoids. Hum Biol 1963 ; 35 : 377-436.
- 2. Sibley CG, Ahlquist JE. The phylogeny of the hominoid primates as indicated by DNA-DNA hybridization. J Mol Evol 1984 ; 20 : 2-15.
- 3. Chou HH, Takematsu H, Diaz S, et al. A mutation in human CMP-sialic hydroxylase occured after the Homo-Pan divergence. Proc Natl Acad Sci USA 1998 ; 95 : 11751-6.
- 4. Satta Y, Klein J, Takahata N. DNA archives and our nearest relative: the trichotomy problem revisited. Mol Phyl Evol 2000 ; 14 : 259-75.
- 5. Chen FC, Li WH. Genomic divergence between humans and other hominoids and the effective population size of the common ancestor of humans and chimpanzees. Am J Hum Genet 2001; 68: 444-56.
- 6. Ebersberger I, Metzler D, Schwarz C, Pääbo S. Genomewide comparison of DNA sequences between humans and chimpanzees. Am J Hum Genet 2002 ; 70 : 1490-7.
- 7. Gilad Y, Man O, Pääbo S, Lancet D. Human specific loss of olfactory receptor genes. Proc Natl Acad Sci USA 2003 ; 100 : 3324-7.
- 8. Hellmann I, Zöllner S, Enard W, et al. Selection on human genes as revealed by comparisons to chimpanzee cDNA. Genome Res 2003 ; 13 : 831-7.
- 9. Sakate R, Osada N, Hida M, et al. Analysis of 5’-end sequences of chimpanzee cDNAs. Genome Res 2003 ; 13 : 1022-6.
- 10. Wildman DE, Uddin M, Liu G, et al. The 99,4 % nonsynonymous and 98,4 % synonymous sequence identity between chimpanzees and humans: implications for Darwinian selection and for enlarging the genus Homo. Proc Natl Acad Sci USA 2003 ; 100 : 7181-8.
- 11. Clark AG, Glanowski S, Nielsen R, et al. Inferring nonneutral evolution from human-chimp-mouse orthologous gene trios. Science 2003 ; 302 : 1960-3.
- 12. Evans PD, Anderson JR, Vallender EJ, et al. Adaptive evolution of ASPM, a major determinant of cerebral cortical size in humans. Hum Mol Genet 2004 ; 13 : 489-94.
- 13. Gilad Y, Wieba V, Przeworski M, et al. Loss of olfactory receptor genes coincides with the acquisition of full trichromatic vision in primates. PLOS Biology 2004 ; 2 : 120-5.
List of figures
Figure 1
Phylogénie du gène ASPM chez les primates.